摘要:冠状病毒的流行对公共卫生构成了重大的威胁,冠状病毒的非结构性蛋白(non-structural protein, NSP)的氨基酸序列、蛋白结构和功能在不同亚型的冠状病毒中高度保守[1]。NSP蛋白对于冠状病毒的免疫拮抗,病毒双层衣壳的组装,核酸的结合,解旋酶活性,RNA序列的校阅等重要的病毒全生命周期的活动都必不可少[2]。其中NSP10可以显著提高NSP14的EXoN区域的活性,从而有效减少RNA病毒的核苷酸错配[3]。另外,NSP10激活和调节NSP16的2’-O-甲基转移酶活性,选择性地转化cap-0成cap-1[4,5]。NSP10蛋白含有两个锌指结构域,其中锌离子结合位点1是由残基C4327,C4330和H4343形成,结合位点2是由残基C4370, C4373, C4381和C4383形成[6]。NSP10锌指结构域的配位残基在多种冠状病毒亚型上100%保守,在病毒复制过程中起重要作用[6]。目前未发现靶向NSP蛋白的小分子抑制剂,应用SPR高通量药物筛选验证针对NSP10锌指结构域虚拟筛选出的化合物后,再用病毒筛选是一个可选择的开发途径。
关键词: Surface plasmon resonance, Non-structural protein, High throughput screening
材料与试剂
- 96孔板封膜 100片装(Cytiva, 产品目录号: 28975816)
- 96孔HTS微孔板 250 μL(Greiner, 产品目录号: 650101)
- 96孔储样板 1 mL(Greiner, 产品目录号: 780201)
- DMSO (Sigma-Aldrich, 产品目录号: D5879-500ML)
- 迷你透析管 10K 0.5 mL 25只装(Thermo, 产品目录号: P00090779)
- NSP10 蛋白(Sino Biological, 产品目录号: 40599-VNCE)
- S系列感应芯片CM5 10片装(Cytiva, 产品目录号: 29149603)
- 10× PBS-P缓冲溶液1,000 mL(Cytiva, 产品目录号: 28995084)
- 醋酸钠 pH 4.0 50 mL(Cytiva, 产品目录号: 14100349)
- 醋酸钠 pH 4.5 50 mL(Cytiva, 产品目录号: 14100350)
- 醋酸钠 pH 5.0 50 mL(Cytiva, 产品目录号: 14100351)
- 醋酸钠 pH5.5 50 mL(Cytiva, 产品目录号: 14100352)
- 氢氧化钠 50 mM(Cytiva, 产品目录号: 14100358)
仪器设备
- Biacore 8K 高通量生物分子相互作用分析筛选系统(Cytiva, GE health, 29337793)
- 高速离心机(Eppendorf centrifuge, 5430)
- 离心机(Eppendorf centrifuge, 5910R)
- 洗板分液机(Thermo, MultiDrop Combi)
- 离心机(IKA, Mini G)
- 涡旋震荡器(上海生工, WH-861)
- 超声仪(昆山舒美, 40KHZ)
- 层析冷柜(永联生物, UL1500)
- 震荡器(其林贝尔, TS-2)
实验步骤
一、实验准备
- 运行Biacore 8K 系统自检(System check),确认微流控系统的精密度在可控范围内。
- 用透析管对蛋白的缓冲液进行透析,在四度冰柜内低速震荡24小时,中间隔8小时换一次1× PBS-P的缓冲溶液。
- 将化合物用DMSO溶解成10 mM的母液,充分震荡后离心。剔除溶解度不佳的化合物,或降低母液浓度至可完全溶解。
- 取10× PBS-P 105 mL,另加入895 mL的双蒸水配置成1,000 mL 1.05× PBS-P,充分震荡后,超声10 min除气泡。另取50 mL 10× PBS-P稀释成 500 mL 1× PBS-P 充分震荡后,超声10 min 除气泡。
- 将维护芯片置换成CM5芯片,同时将进缓冲液的管路换到1× PBS-P的缓冲溶液内,并运行置换缓冲溶液的程序一次。
二、偶联配体
本实验采用氨基偶联法,考虑蛋白和分析物分子量的比例在100内,选用CM5 芯片。选用1× PBS-P缓冲溶液运行实验程序。
- 点击New Method, 选择Surface Preparation中的pH scouting。将蛋白样品用不同pH的醋酸钠缓冲溶液(pH 5.5、5.0、4.5、4.0) 稀释到1 μg/mL。运行pH scouting的程序,预富集结果显示pH 4.0为优选条件,具体见结果与分析的图2。
- 点击New Method, 选择Surface preparation, 选择Immobilization, Chip type 选择CM5。使用1通道做氨基偶联测试,将用于封闭通道的乙醇胺替换成运行缓冲溶液1× PBS-P (进样时间420 s, 10 μL/min)。取1 μg/μL蛋白5 μL稀释在135 μL pH 4.0的醋酸钠溶液里(35.7 μg/ mL),进样时间为1,200 s, 流速设置为5 μL/min。 EDC.HCl (55 μL)和NHS(55 μL)预混后做活化试剂(进样时间420 s, 10 μL/min)。结果显示偶联量有10,000 RU以上,具体见结果与分析中图3。故采用相同方法偶联其他通道,并用乙醇胺做通道封闭。偶联结果显示其他通道也有10,000 RU以上的偶联量,具体见结果与分析中图4和表1。运行置换缓冲溶液的程序三次,部分并非稳定偶联在芯片上的蛋白会从芯片上脱离。
三、小分子化合物单浓度高通量筛选
- 溶剂矫正液配制:取19 mL 1.05× PBS-P, 加入0.9 mL DMSO,配制成4.5% DMSO PBS-P缓冲溶液,记为溶剂矫正液1。取19 mL 1.05× PBS-P, 加入1.16 mL DMSO,配制成5.8% DMSO PBS-P缓冲溶液,记为溶剂矫正液4。用于配制缓冲溶液的DMSO在使用和保存过程中注意密封,避免吸湿。溶剂矫正液1和溶剂矫正液4充分震荡后,超声10 min,有利于提高溶液的均一性。取7 mL 溶剂校正液1,与3 mL 溶剂校正液充分混合,得到溶剂校正液2。取 3 mL 溶剂校正液1,与7 mL 溶剂校正液充分混合,得到溶剂校正液3。
- 小分子样品的准备:将9.5 mL 1.05× PBS-P溶液与40 mL 含5% DMSO 的PBS-P运行缓冲液充分混合后,用8通道的排枪取198 μL加入0.2 mL的联排管里,将2 μL 10 mM DMSO溶解的小分子母液依次加入编号后的联排管中,充分混合震荡,离心20 min去除震荡时产生的微小气泡(离心机 IKA mini G)。
- 在Biacore 8K 的控制软件上新建程序,选择多循环动力学,结合时长设置为180 s, 解离时长设置为120 s, 流速设置为30 μL/min。可增加Startup便于稳定基线,本实验Startup设置了五个循环。另外本实验每个化合物用含5% DMSO 的1× PBS-P运行缓冲溶液作为两个零浓度,如化合物的数目较多,通道有限的情况下可以设置一个零浓度。选用含5% DMSO 的1× PBS-P缓冲溶液运行实验程序。
四、小分子化合物亲和力测定
- 根据单浓度高通量筛选结果的响应情况,去除阴性结果的化合物,剩下化合物按响应值大小排序。优先测试响应值较高的化合物。参照先前的方法配制100 μM的样品液 400 μL,取出200 μL 2倍梯度稀释依次产生50 μM,25 μM,12.5 μM,6.25 μM,3.125 μM,1.5625 μM,0.78125 μM溶液各200 μL。一般虚筛所得化合物的结合力多为微摩尔级别,故如阳性化合物较多的情况下,最后两个低浓度也可不用做。溶剂矫正液的配制方法同前。
- 程序方法和先前单浓度相似,选择多循环动力学,结合时间设置为180 s, 解离时间设置为120 s, 流速设置为30 μL/min,Startup设置为五个循环。每个化合物用含5% DMSO 的1× PBS-P缓冲溶液设置三个零浓度,先运行两个零浓度循环,再运行由低到高梯度稀释的化合物溶液循环,最后运行一个零浓度循环。化合物在零浓度下的传感曲线偶尔稳定性欠佳,有多个可选零浓度可提高扣减后曲线的平稳性。具体见图1。选用含5% DMSO 的1× PBS-P缓冲溶液运行实验程序。

图1 采用多循环动力学测试化合物的结合力的设置方法
结果与分析
配体偶联pH scouting结果:如图2所示,通道 4(pH 4.0 醋酸钠溶液)蛋白富集在芯片上的量最多,富集曲线(37 s 至 217 s)的上升速度明显高于其他通道,因此可以判定pH 4.0的醋酸钠溶液是较优的蛋白静电富集条件。
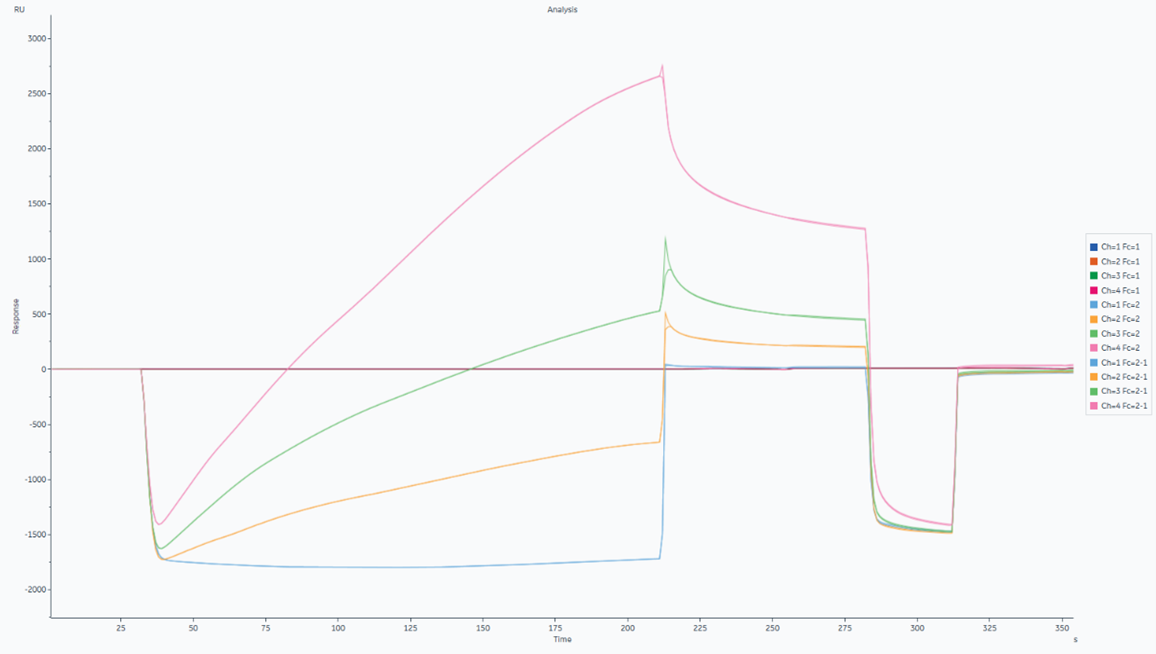
图2 配体偶联pH scouting的信号传感图
配体偶联Immobilization 结果:图3和图4分别为通道1和其余通道的蛋白固定结果,所有通道的蛋白固定值都可以达到10,000RU(见表1)。
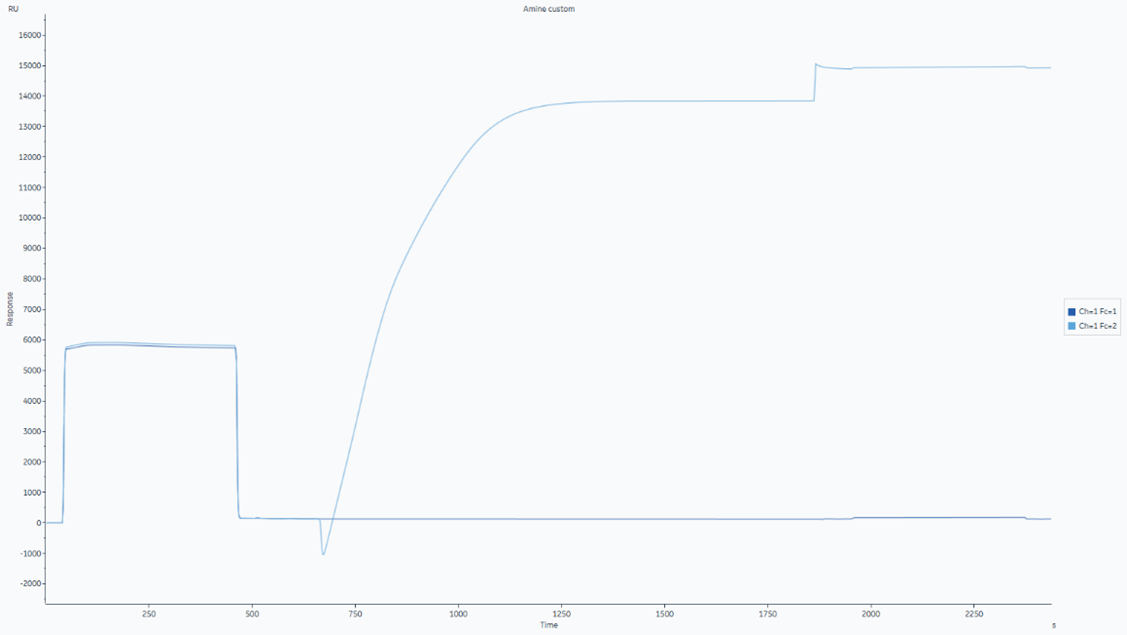
图3 通道1配体偶联Immobilization的信号传感图
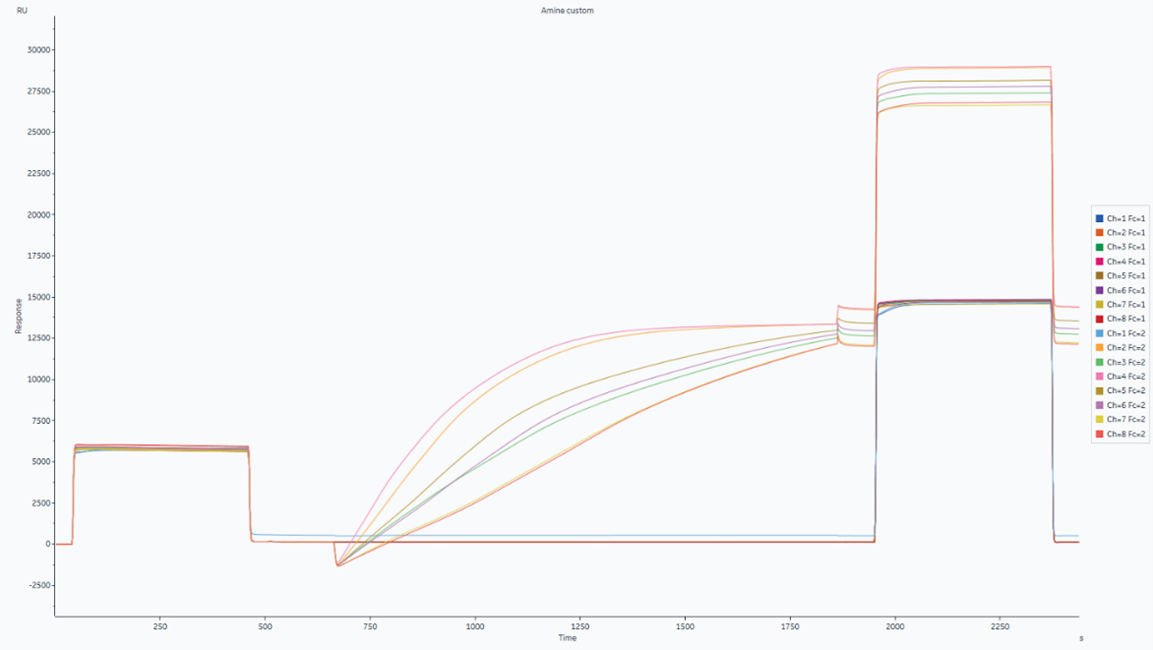
图4 通道2–8配体偶联Immobilization的信号传感图
表1 通道1-8蛋白配体偶联的最终响应值
| Channel | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 |
| Response Final (RU) | 1,4920 | 14381 | 12759 | 1,4404 | 1,3550 | 1,3089 | 1,2233 | 1,2146 |
小分子化合物单浓度高通量筛选:分析方法选择binding screen中的LMW screening-Evaluation method。如图5所示化合物43有较高的响应值,将作为初筛出的阳性药物进行下一轮不同浓度梯度的多循环动力学筛选。如图6所示化合物96响应值较低,将不作为下一轮筛选的样品。
 图 5 化合物43单浓度100 μM筛选的信号传感图
图 5 化合物43单浓度100 μM筛选的信号传感图 图6 化合物96 单浓度100 μM筛选的信号传感图
图6 化合物96 单浓度100 μM筛选的信号传感图小分子化合物多浓度梯度的筛选:分析方法选择Multi-cycle kinetics/affinity,检查BlanK一栏,去除波动较大的零浓度, Kinetic fit model按默认选择1:1,Affinity fit model按默认选择Steady state affinity。Initial values将Kinetics中的Rmax、tc、RI和Drift调整为Fit local,提高动力学拟合曲线质量。拟合结果示意图见图7,化合物43能够浓度依耐地结合到NSP10蛋白上,结合力KD为3.83 × 10-5 M。

图7 化合物43 多浓度梯度的信号拟合图
实验注意要点
- 用SPR筛选小分子药物时,由于大多数情况下小分子化合物在运行缓冲溶液中的溶解性有限,因此一般多需要加入一定比例的DMSO助溶。加入DMSO的比例根据小分子的溶解性调整。本实验中虚筛所得的多种不同骨架的小分子,溶解性差异较大,因此实验中DMSO的添加量调整到5%。由于DMSO的折光率较高,如果配置的运行缓冲溶液时DMSO的添加量不够准确,或者配置最高浓度的小分子样品时DMSO的浓度不够准确,极易造成后续溶剂校正的失败。推荐实验前用制备运行缓冲溶液的DMSO样品溶解小分子样品,不建议使用存放时间过长的小分子DMSO母液。
- 测试不同类型的小分子药物前,建议和对方沟通好化合物的水溶性,对于溶解性较差的化合物,可以制成含5% DMSO的最高浓度的测试样品,并观察测试样品是否有肉眼可见的浑浊,放置过夜后显微镜下能否看到晶体。如果化合物在最高浓度的测试样本里不能完全溶解,可使用更低浓度的小分子母液保证能够完全溶解。保证最高浓度的测试样品的均一才能得到可靠稳定的实验结果。
致谢
感谢国家自然科学基金国际(地区)合作研究项目82061138003对项目的资助。
参考文献
- V'kovski, P., Gerber, M., Kelly, J., Pfaender, S., Ebert, N., Braga Lagache, S., Simillion, C., Portmann, J., Stalder, H., Gaschen, V., et al. (2019). Determination of host proteins composing the microenvironment of coronavirus replicase complexes by proximity-labeling. eLife. 8: e42037. https://doi.org/10.7554/elife.42037
- Bouvet, M., Lugari, A., Posthuma, C. C., Zevenhoven, J. C., Bernard, S., Betzi, S., Imbert, I., Canard, B., Guillemot, J. C., Lécine, P., et al. (2014). Coronavirus Nsp10, a Critical Co-factor for Activation of Multiple Replicative Enzymes. J Biol Chem. 289(37): 25783–25796. https://doi.org/10.1074/jbc.m114.577353
- Chen, Y., Cai, H., Pan, J., Xiang, N., Tien, P., Ahola, T. and Guo, D. (2009). Functional screen reveals SARS coronavirus nonstructural protein nsp14 as a novel cap N7 methyltransferase. Proc Natl Acad Sci USA. 106(9): 3484–3489. https://doi.org/10.1073/pnas.0808790106
- Bouvet, M., Debarnot, C., Imbert, I., Selisko, B., Snijder, E. J., Canard, B. and Decroly, E. (2010). In Vitro Reconstitution of SARS-Coronavirus mRNA Cap Methylation. PLoS Pathog. 6(4): e1000863. https://doi.org/10.1371/journal.ppat.1000863
- Lugari, A., Betzi, S., Decroly, E., Bonnaud, E., Hermant, A., Guillemot, J. C., Debarnot, C., Borg, J. P., Bouvet, M., Canard, B., et al. (2010). Molecular Mapping of the RNA Cap 2′-O-Methyltransferase Activation Interface between Severe Acute Respiratory Syndrome Coronavirus nsp10 and nsp16*. J Biol Chem. 285(43): 33230–33241. https://doi.org/10.1074/jbc.m110.120014
- Rosas-Lemus, M., Minasov, G., Shuvalova, L., Inniss, N. L., Kiryukhina, O., Brunzelle, J. and Satchell, K. J. F. (2020). High-resolution structures of the SARS-CoV-2 2′- O -methyltransferase reveal strategies for structure-based inhibitor design. Sci Signaling. 13(651): eabe1202. https://doi.org/10.1126/scisignal.abe1202
Copyright: © 2023 The Authors; exclusive licensee Bio-protocol LLC.
引用格式:李云齐, 陈彦文, 谭云. (2023). 应用SPR技术验证结合NSP10的小分子. // Surface Plasmon Resonance (SPR) Protocol eBook.
Bio-101: e1011005. DOI:
10.21769/BioProtoc.1011005.
 How to cite:
How to cite: Li, Y. Q., Chen, Y. W. and Tan, Y. (2023). Biophysical Evaluation of Small Molecules Potentially Targeting NSP10 by Surface Plasmon Resonance. // Surface Plasmon Resonance (SPR) Protocol eBook.
Bio-101: e1011005. DOI:
10.21769/BioProtoc.1011005.
