材料与试剂
- 重组SARS-CoV-2 RBD蛋白 (Sino Biological, 产品目录号: 40592-V08H)
- hACE2 蛋白 (Sino Biological, 产品目录号: 10108-H08H)
- 小分子药物库 (FDA-approved Drug Library, TargetMol, 产品目录号: L4200)
- S系列CM5芯片 (Cytiva, Series S Sensor Chip CM5, 产品目录号: 29149603)
- 氨基偶联试剂盒 (Cytiva, 产品目录号: BR100050)
- 醋酸钠缓冲液 (Cytiva, Acetate 4.5, 产品目录号: BR100350; Acetate 5.0, 产品目录号: BR100351; Acetate 5.5, 产品目录号: BR100352)
- 96孔板 (Cytiva, 产品目录号: BR100503)
- 96孔板封膜 (Cytiva, 产品目录号: 28975816)
- 10× PBS-P缓冲液 (Cytiva, 产品目录号: 8995084)
- 二甲基亚砜 (DMSO, MP, 产品目录号: 196055)
- 50 mM NaOH溶液 (Cytiva, 产品目录号: BR100358)
- 1.5 mL EP管 (KIRGEN, 产品目录号: KG2211)
- 1000 mL 0.2 μm NY过滤器 (康宁, 产品目录号: 430515)
- 1.05× PBS-P缓冲液 (见溶液配方)
- 含5% DMSO的1× PBS-P缓冲液 (见溶液配方)
- 蛋白母液 (见溶液配方)
- 小分子药物母液 (见溶液配方)
- 不同浓度的DMSO溶剂校正溶液 (见溶液配方)
- 50% DMSO溶液 (见溶液配方)
- 5 mM NaOH溶液 (见溶液配方)
仪器设备
- Milli-Q纯水仪 (密理博, millipore)
- 表面等离子共振仪 (Cytiva, Biacore 8K)
- 台式离心机 (贝克曼, Microfuge 20)
- 循环水多用真空泵 (郑州特尔仪器设备有限公司, SHZ-D(III))
实验步骤
一、实验准备
打开Biacore 8K仪器和控制软件Biacore 8K Control Software,输入软件登录密码。流动池和样品舱的温度在4–40 °C可调,通常都设置为25 ℃。
将仪器右侧标记buffer的管子插入1.05× PBS-P缓冲液瓶子里,将标记water和reagent的管子插入超纯水的瓶子里。
在Biacore 8K Control Software的System setup模块,点击Change chip,放入新的CM5芯片,在仪器控制软件上填写芯片相关信息,然后点击Dock chip。
在Biacore 8K Control Software的System setup模块,点击Change solutions,默认设置,点击Ready to start。缓冲液会以较高的流速冲洗仪器整个内部流路系统,整个过程耗时6分钟。
二、配体偶联
(1)计算配体偶联量
根据以下公式计算配体目标偶联量:

其中,Rmax为芯片表面最大结合容量,通常设为100 RU;Analyte MW为小分子分子量,ligand MW为蛋白分子量;Sm为化学计量比,通常设成1;配体目标偶联量通常设为1.5倍的RL。在本研究案例中,SARS-CoV-2 RBD的分子量为26 kDa,hACE2的分子量为86 kDa,小分子药物的分子量范围是300~1,900 Da。经公式计算,SARS-CoV-2 RBD目标偶联量为2,000~14,000 RU,hACE2目标偶联量为6,700–43,000 RU。
(2)配体预富集
将SARS-CoV-2 RBD蛋白和hACE2蛋白粉末用超纯水溶成1 mg/mL母液。
根据蛋白等电点(SARS-CoV-2 RBD等电点为8.9,hACE2等电点为5.5),使用10 mM pH 5.5和10 mM pH 5.0的醋酸钠溶液稀释SARS-CoV-2 RBD蛋白至20 μg/mL,使用10 mM pH 5.0和4.5的醋酸钠溶液稀释hACE2蛋白至20 μg/mL。
在Biacore 8K Control Software里,点击New method,选择Surface preparation,打开pH scouting方法。方法里包含四个步骤。第一步Method definition,设置Analyte的Contact time为120 s;第二步Variables and positioning,选择channel 1~4,修改Analyte Solution名称分别为SARS-CoV-2 RBD acetate pH 5.5、SARS-CoV-2 RBD acetate pH 5.0、hACE2 acetate pH 5.0、hACE2 acetate pH 4.5;第三步Cycle overview不需要做任何改动;第四步Plate layout,显示了96孔板里样品放置位置和需要体积(图1)。按照第四步的要求将样品加到96孔板里。考虑到进样针吸取样品时会有死体积,因此每个样品多加10 µL体积。

图1 配体预富集实验方法编辑
在Biacore 8K Control Software的Instrument control界面,Hotel door处点击open,把样品架从样品舱里取出来,把加好样品的96孔板放置到样品架上,然后放到仪器里面,把样品舱的门关闭。在Methods界面,点击第四步Plate layout后面的Send to queue,点击Ready to start和Save,方法开始运行。
根据预富集实验结果(图2),确定SARS-CoV-2 RBD蛋白的偶联条件为10 mM pH 5.5的醋酸钠溶液,hACE2蛋白的偶联条件为10 mM pH 4.5的醋酸钠溶液。

图2 预富集实验结果
(3)配体偶联
在Biacore 8K Control Software里,点击New method,选择Surface preparation,打开Immobilization方法。方法里包含两个步骤。如图3,第一步Method definition,选择Immobilize in Fc 2, activate/deactivate in 1,点击Enter custom mode。将EDC+NHS的Contact time设置成1200 s,Ligand的Contact time设置成600 s,Ethanolamine的Contact time设置成1200 s。Channel选择1–8。其中Channel 1–4固定SARS-CoV-2 RBD蛋白,Channel 5–8固定hACE2蛋白。第二步Positioning and plate layout,显示了96孔板里样品放置位置和需要体积(图4),按照要求将样品加到96孔板里。考虑到进样针吸取样品时会有死体积,因此每个样品多加10 µL体积。其中EDC、NHS和Ethanolamine用氨基偶联试剂盒的配套试剂,SARS-CoV-2 RBD蛋白用10 mM pH 5.5的醋酸钠稀释至20 μg/mL,hACE2蛋白用10 mM pH 4.5的醋酸钠稀释至40 μg/mL。

图3 配体偶联的方法设置

图4 配体偶联的样品板位置和所需样品体积
在Biacore 8K Control Software的Instrument control界面,在Hotel door处点击open,把样品架从样品舱里取出来,把加好样品的96孔板放置到样品架上,然后放到仪器里,关闭样品舱的门。在Methods界面,点击第二步Positioning and plate layout后面的Send to queue,点击Ready to start和Save,方法开始运行。
运行结束后,结果显示SARS-CoV-2 RBD蛋白偶联了~ 14,000 RU(图5),hACE2蛋白偶联了~10,000 RU(图6)。

图5 SARS-CoV-2 RBD蛋白偶联情况

图6 hACE2蛋白偶联情况
三、新型冠状病毒小分子抑制剂的筛选
前期研究工作中,通过虚拟筛选确定了20个小分子药物(见表1)可能作为潜在的小分子抑制剂,抑制SARS-CoV-2 RBD和hACE2结合。
表1 通过虚拟筛选确定的20个小分子药物信息
| 序号 | 小分子药物名称 | Cas号 | 分子量 (Da) |
| 1 | Tannic acid | 1401–55–4 | 1701 |
| 2 | Nystatin | 1400–61–9 | 926 |
| 3 | Sodium aescinate | 20977–05–3 | 1124 |
| 4 | Teichomycin | 61036–62–2 | 1879 |
| 5 | Aescin | 6805–41–0 | 1131 |
| 6 | Ginsenoside Rc | 11021–14–0 | 1079 |
| 7 | Amphotericin B | 1397–89–3 | 924 |
| 8 | Acarbose | 56180–94–0 | 645 |
| 9 | Geniposide | 24512–63–8 | 388 |
| 10 | Vancomycin HCL | 1404–93–9 | 1485 |
| 11 | Mevastatin | 73573–88–3 | 390 |
| 12 | Ambroxol | 18683–91–5 | 378 |
| 13 | Dapagliflozin | 461432–26–8 | 408 |
| 14 | Canagliflozin | 842133–18–0 | 444 |
| 15 | Simvastatin | 79902–63–9 | 418 |
| 16 | Cytomel | 55–06–1 | 672 |
| 17 | Sodium Picosulfate | 10040–45–6 | 481 |
| 18 | Fasiglifam | 1000413–72–8 | 524 |
| 19 | Pseudolaric acid B | 82508–31–4 | 432 |
| 20 | Thyroxine | 51–48–9 | 776 |
接下来,使用Biacore 8K对这20个小分子药物与SARS-CoV-2 RBD和hACE2的亲和力和动力学进行了检测。
将仪器右侧1.05× PBS-P缓冲液换成含5% DMSO的1×PBS-P缓冲液,在Biacore 8K Control Software的System setup模块,点击Change solutions,默认设置,点击Ready to start。
在1.5 mL EP管里,先加入9 µL DMSO,再加入6 µL 10 mM小分子药物母液,混合后加入285 µL 1.05× PBS-P溶液,混合均匀即得到200 µM 5% DMSO的小分子药物溶液。用离心机13,000× g离心5分钟,然后取出240 µL上清液作为小分子药物最高浓度。接下来,用含5% DMSO的1× PBS-P配置小分子药物浓度梯度,2倍稀释,共九个浓度梯度。
在Biacore 8K Control Software,点击New method,选择Kinetics/affinity,在LMW里选择LMW kinetics/affinity,打开LMW multi-cycle kinetics/affinity方法。第一步Method definition,将Concentration unit改成µM。在Analysis里,Analyte的Contact time设置成60 s,Dissociation time设置成90 s。将Startup里的Analyte设置成和Analysis里完全一样的实验条件。第二步Variables and positioning,在Analysis里,Add cycle至30个循环,将Analyte Solution改为小分子药物名称,浓度分别设置成0、0.78、1.56、3.13、6.25、12.5、25、50、100、200 µM。在Startup里,Add cycle至5个循环。第三步Cycle overview不需要做任何改动。第四步Plate layout,如图7–10所示,将样品配好加到96孔板里,其中0浓度和buffer为含5% DMSO的1× PBS-P缓冲液,Solvent Correction 1–4为表3里的四点溶剂校正溶液。然后给96孔板贴上封膜,放到仪器里,点击 Send to queue,Ready to start和Save,方法开始运行。
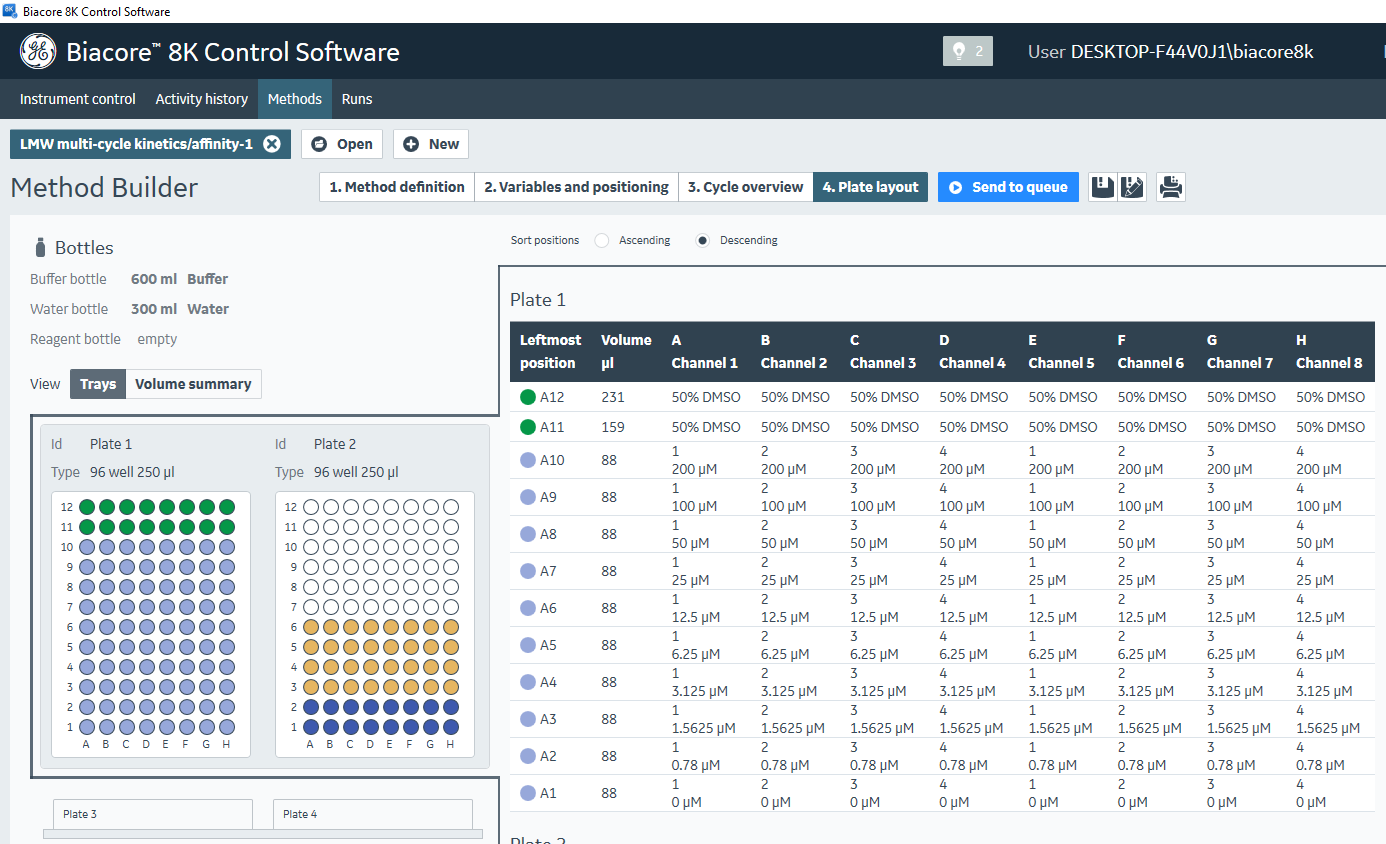
图7 Plate 1的样品名称、位置和所需体积

图8 Plate 2的样品名称、位置和所需体积

图9 Plate 3的样品名称、位置和所需体积

图10 Plate 4的样品名称、位置和所需体积
由于Channel 1~4固定SARS-CoV-2 RBD蛋白,Channel 5~8固定hACE2蛋白,使用Biacore 8K仪器一次最多放置4块96孔板,因此每个蛋白一轮最多可以做12个化合物。在本实验案例中,20个小分子药物需要使用Biacore 8K仪器做两轮实验。
四、小分子药物对SARS-CoV-2 RBD和hACE2相互作用的竞争抑制研究
通过上述Biacore 8K的结合实验,确定了四种小分子药物Tannic acid、Amphotericin B、Aescin和Fasigifam对SARS-CoV-2 RBD或hACE2的亲和力较强。接下来使用Biacore 8K独特的A-B-A进样方法研究这四种小分子药物对SARS-CoV-2 RBD和hACE2相互作用的竞争抑制作用。
用一张新的CM5芯片,通过上述配体偶联方法固定hACE2,偶连量为~2,000 RU。Solution A为单独的小分子药物溶液,用来预先和芯片上的hACE2孵育。小分子药物+SARS-CoV-2 RBD作为Solution B。通过观察加入小分子药物后,SARS-CoV-2 RBD与hACE2的结合信号是否降低来判断小分子药物对SARS-CoV-2 RBD和hACE2相互作用的竞争抑制作用。
在Biacore 8K Control Software里,点击New method,在Assay Development里选择Buffer scouting using A-B-A。第一步Method definition,如图11,在Analysis里,Injections A的Pre-analyte和Post-analyte的contact time设置成60 s,Injections B的Contact time设置成120 s,Concentration为40 nM。Regeneration使用5 mM NaOH溶液,Contact time设置成30 s,Flow rate为30 µL/min。第二步Variables and positioning,Channels选择1~4,Solution A为小分子药物,浓度为100 µM,Solution B为小分子药物+SARS-CoV-2 RBD,其中SARS-CoV-2 RBD的浓度为40 nM,小分子药物浓度为100 µM。Solution B中,只加SARS-CoV-2 RBD,不加小分子药物,作为对照。第三步Cycle overview不需要做任何改动。第四步Plate layout,如图12,将样品配好加到96孔板里,贴上封膜,放到仪器里,点击Send to queue,Ready to start和Save,方法开始运行。

图11 A-B-A实验的方法设置

图12 样品名称、位置和所需体积
进一步,使用Biacore 8K的A-B-A方法研究小分子药物Tannic acid抑制SARS-CoV-2 RBD和hACE2相互作用的量效关系。在A-B-A的第二步Variables and positioning,Solution B设置为不同浓度的小分子药物+40 nM SARS-CoV-2 RBD,Solution A为不同浓度的小分子药物。图13~15为样品板里的样品名称、位置和所需体积。

图13 Plate 1的样品名称、位置和所需体积

图14 Plate 2的样品名称、位置和所需体积

图15 Plate 3的样品名称、位置和所需体积
实验注意要点
- 配体偶联:
对于蛋白和小分子相互作用实验,配体蛋白偶联量约10,000 RU即可开展实验。如果不确定配体蛋白的实际偶联效果,可以在Immobilization方法里先去掉封闭Ethanolamine这步,等蛋白偶联量达到预期后,再使用Immobilization方法单独运行Ethanolamine这步。 - 小分子药物溶液配制:很多小分子药物溶解性不好,需要DMSO助溶。小分子药物配成最高实验浓度后,需要离心观察是否有沉淀,如果离心后没有看到沉淀,则取上清液做浓度梯度稀释,如果离心后看到沉淀,则需要降低小分子药物的最高实验浓度。在表面等离子共振仪实验中,小分子药物做分析物,其浓度必须是准确的,否则会影响亲和力KD的数值。
- 小分子药物A-B-A竞争抑制实验的浓度设置:
Solution A为小分子药物,如果小分子药物和芯片上的蛋白有一定的结合信号,小分子药物的浓度需要设置为能使芯片上蛋白位点饱和的浓度,数值通常比较高,如100 µM或200 µM,并且小分子药物的结合信号可以通过设置A-A-A作为零浓度进行扣减。
Solution B中蛋白的浓度通常设为蛋白和蛋白亲和力KD同样数量级的浓度。
由于很多小分子药物是快结合快解离的模式,因此Solution B中需要同样添加和Solution A中一样浓度的小分子药物。
结果与数据分析
(1)小分子药物与SARS-CoV-2 RBD的亲和力和动力学检测:
使用Biacore 8K的分析软件Biacore Insight Evaluation software对结果进行分析。在Create new evaluation界面,找到要处理的文件,在Predefined界面,选择Kinetics/affinity里的LMW,选择LMW multi-cycle kinetics-Evaluation method。在溶剂校正界面,点击Apply and close。仪器默认的会把进样前后的部分曲线截掉,如果不想截掉,点击图右侧的小齿轮,在Remove range里,去掉勾选的Remove ranges at start and end of injections。在Select sensorgrams,点击More option,在Analyte里选择浓度。在Kinetics/Affinity mode点击Kinetics,Fit models选择1:1 binding模型,在Initial values里,RI选择Fit local,Rmax选择Fit local,点击Perform fit,即得到拟合结果,包括结合速率ka,解离速率kd,Rmax和亲和力KD。
如图16,结果显示有15个小分子药物与SARS-CoV-2 RBD有特异性结合,亲和力范围是179 nM到273 µM。其中,Tannic acid、Amphotericin B和Aescin与SARS-CoV-2 RBD的亲和力最强,并且具有较慢的解离速率,提示可能具有更长的滞靶时间,因此用来做进一步的竞争抑制实验。

图16 Biacore 8k检测小分子药物与SARS-CoV-2 RBD的亲和力和动力学。
(2)小分子药物与hACE2的亲和力和动力学检测:
使用Biacore 8K的分析软件Biacore Insight Evaluation software对结果进行分析。选择LMW multi-cycle kinetics-Evaluation method,在溶剂校正界面,点击Apply and close,在Kinetics/Affinity mode位置点击Kinetics,Fit models选择1:1 binding模型,在Initial values里,RI选择Fit local,Rmax选择Fit local,点击Perform fit,即得到拟合结果,包括结合速率ka,解离速率kd,Rmax和亲和力KD。
如图17,结果显示有9个小分子药物与hACE2有特异性结合,亲和力范围是8.55 µM到250 µM。其中,Tannic acid、Amphotericin B和Fasigifam与hACE2的亲和力最强,用来做进一步的竞争抑制实验。

图17 Biacore 8k检测小分子药物与hACE2的亲和力和动力学。
(3)小分子药物对SARS-CoV-2 RBD和hACE2相互作用的竞争抑制研究
使用Biacore 8K的A-B-A进样方法研究Tannic acid、Amphotericin B、Aescin和Fasigifam四种小分子药物能否对SARS-CoV-2 RBD和hACE2的结合发挥竞争抑制作用。如图18,SARS-CoV-2 RBD和hACE2本身存在较强的相互作用(蓝线),在100 µM Tannic acid作用下,SARS-CoV-2 RBD和hACE2几乎不结合,而在100 µM Amphotericin B、Aescin或Fasigifam作用下,SARS-CoV-2 RBD与hACE2的结合信号没有明显下降,即没有表现出明显的竞争抑制作用。
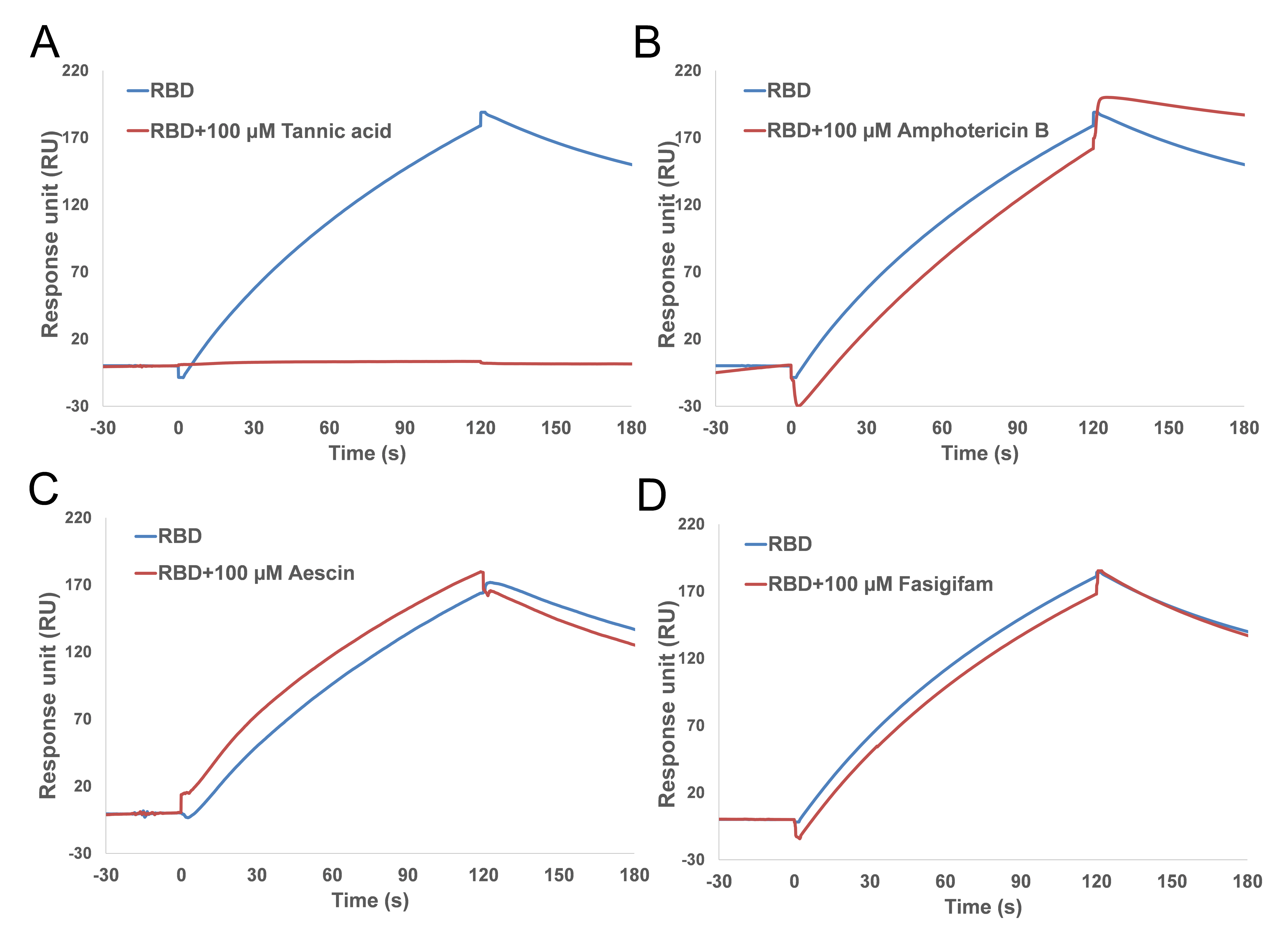
图18 小分子药物对SARS-CoV-2 RBD和hACE2结合的竞争抑制研究。蓝线:SARS-CoV-2 RBD和hACE2的结合信号。红线:加入小分子药物后,SARS-CoV-2 RBD和hACE2的结合信号。
进一步,如图19,研究了不同浓度的Tannic acid对SARS-CoV-2 RBD与hACE2结合的影响。可以看到,随着Tannic acid浓度升高,SARS-CoV-2 RBD与hACE2的结合信号越来越低,当Tannic acid浓度达到25 µM时,已完全抑制SARS-CoV-2 RBD与hACE2的结合,从体外分子水平证明了Tannic acid能作为新型冠状病毒的潜在的小分子抑制剂直接阻断SARS-CoV-2 RBD与hACE2的结合。

图19 不同浓度的Tannic acid对SARS-CoV-2 RBD与hACE2结合的影响
溶液配方
- 1.05× PBS-P缓冲液
量取210 mL 10× PBS-P缓冲液,加1,790 mL超纯水,使用0.2 µm过滤器和循环水多用真空泵过滤到装缓冲液的瓶子里。 - 含5% DMSO的1× PBS-P缓冲液
从1.05× PBS-P缓冲液中量取1,425 mL到装缓冲液的瓶子里,加入75 mL用0.22 µm有机滤膜过滤后的DMSO。 - 蛋白母液
将SARS-CoV-2 RBD蛋白和hACE2蛋白粉末用超纯水溶成1 mg/mL母液。 - 小分子药物母液
将小分子药物粉末用DMSO溶解并配成10 mM母液。 - 不同浓度的DMSO溶剂校正溶液
按表2,配置4.5% DMSO和5.8% DMSO的PBS-P溶液。
表2 溶剂校正的溶液配方
| 4.5% DMSO | 5.8% DMSO |
| 1.05× PBS-P | 9.5 mL | 9.5 mL |
| 100% DMSO | 0.45 mL | 0.58 mL |
| 终体积 | ~10 mL | ~10 mL |
4.5%和5.8% DMSO的PBS-P溶液按一定比例混合得到四点溶剂校正溶液(表3)。
表3 四点溶剂校正溶液配方
| 1 | 2 | 3 | 4 |
| 4.5% DMSO | 0 | 400 µL | 2,400 µL | 2,800 µL |
| 5.8% DMSO | 2,800 µL | 2,400 µL | 400 µL | 0 |
- 50% DMSO溶液
取5 mL超纯水,加入5 mL用0.22 µm有机滤膜过滤后的DMSO。 - 5 mM NaOH溶液
取100 µL 50 mM NaOH,加入900 µL超纯水,混匀。
致谢
感谢北京大学药学院天然药物及仿生药物全国重点实验室提供仪器技术支撑。感谢北京大学医学部基础医学院王月丹教授和初明副教授课题组提供实验数据。感谢王子元同学的帮助。
参考文献
- Chen, X., Wang, Z., Wang, J., Yao, Y., Wang, Q., Huang, J., Xiang, X., Zhou, Y., Xue, Y., Li, Y., et al. (2022). Role of tannic acid against SARS-cov-2 cell entry by targeting the interface region between S-protein-RBD and human ACE2. Front Pharmacol. 13: e940628. https://doi.org/10.3389/fphar.2022.940628
引用格式:王静. (2024). 新型冠状病毒小分子抑制剂的筛选和竞争抑制研究. // Surface Plasmon Resonance (SPR) Protocol eBook.
Bio-101: e1011009. DOI:
10.21769/BioProtoc.1011009.
 How to cite:
How to cite: Wang, J. (2024). Screening and Competitive Inhibition Study of Small Molecular Inhibitors against SARS-CoV-2. // Surface Plasmon Resonance (SPR) Protocol eBook.
Bio-101: e1011009. DOI:
10.21769/BioProtoc.1011009.
