摘要: 蛋白与小分子相互作用的研究方法多样,但利用基于表面等离子共振(SPR)技术的 Biacore T200 进行互作研究的方法报道甚少。本文以人血清白蛋白(Human Serum Albumin,简称HSA)和小分子法华林(Warfarin)的相互作用为例,详细描述了分子间相互作用的整个实验流程,包括实验前芯片、缓冲液的准备、HSA蛋白最佳偶联pH值的筛选、偶联过程、分析物Warfarin的芯片表面测试和再生条件的选择、HSA蛋白与Warfarin相互作用时多循环动力学测试、数据分析、实验结束后仪器的处理;同时,还对实验中出现的各种常见问题进行了剖析并提出相应的解决方案,包括仪器状态、传感芯片的选择、对偶联蛋白以及偶联量的要求、缓冲液的选择、非特异吸附的问题、再生试剂的选择以及再生效果的要求、数据拟合质量评价等。本文可为使用SPR技术研究蛋白与小分子化合物相互作用提供详实的参考。
关键词: 表面等离子共振技术, 蛋白与小分子化合物的相互作用, 人血清白蛋白, 法华林, 方法和解决方案
材料与试剂
- 人血清白蛋白(Human Serum Albumin,HSA)(Sigma-Aldrich,产品目录号:MFCD00081418)
- Warfarin (MedChemExpress,产品目录号:HY-B0687)
- Series S CM5芯片(Cytiva,产品目录号: BR-1005-30)
- 氨基偶联试剂盒(Cytiva,产品目录号: BR-1000-50)
- 10× HBS-EP+缓冲液(Cytiva,产品目录号: BR-1006-69)
- 醋酸钠缓冲液 (Cytiva, Acetate 4.0, 产品目录号: BR100349; Cytiva, Acetate 4.5, 产品目录号: BR100350; Acetate 5.0, 产品目录号: BR100351; Acetate 5.5, 产品目录号: BR100352)
- 50 mM NaOH溶液 (Cytiva,产品目录号: BR100358)
- 二甲基亚砜(Sigma,产品目录号: D-8418-50ML)
- 10× HBS-N缓冲液(Cytiva,产品目录号: BR-1006-70)
- BIAtest Solution with HBS-N (Cytiva,产品目录号: 210310)
- 1.5 mL 离心管(赛默飞QSP,产品目录号: 509-GRD-Q)
仪器设备
- 表面等离子共振仪(Cytiva, Biacore T200)
- Milli-Q纯水仪(Millipore,Milli-Q Integral Integral)
软件
- 运行软件: T200 Control Software(Cytiva, Version 3.2)
- 数据处理软件:Evaluation Software(Cytiva, Version 3.2)
实验步骤
一、实验前准备
芯片的准备 选择Tools菜单中Eject Chip,芯片舱门打开,将维护芯片(Maintenance Chip)取出,将CM5芯片按箭头方向放入卡槽,合上舱门;在Chip Type下拉菜单中选择对应的芯片种类,在Chip ID中填入和芯片相关的信息,如果是已经使用过的芯片,选择Reuse Chip,并在Chip ID下拉菜单中找到与之相对应的芯片信息;点击Dock Chip按钮,芯片装载后系统将自动进入Standby状态(图1)。
缓冲液的准备 将10× HBS-EP+ Buffer用超纯水稀释至1× HBS-EP+,放置于仪器托盘左侧,右侧放超纯水。芯片、缓冲液准备完成后,点击tools、more tools、执行Prime功能(图2)。
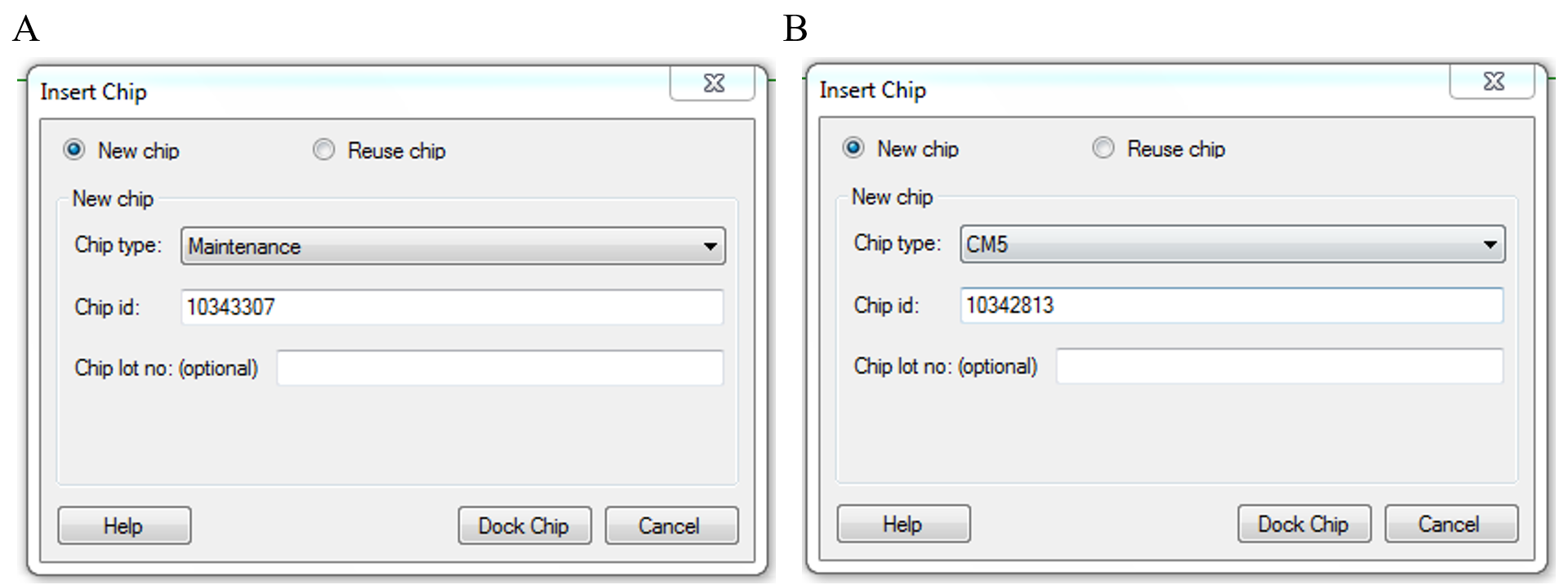
图1 将维护芯片换为CM5芯片。
图2 换芯片、缓冲液后对仪器进行Prime。
二、实验流程
- HSA蛋白偶联pH值的筛选
选用不同pH的醋酸钠缓冲液(10 mM sodium acetate,pH5.5、5.0、4.5、4.0),将HSA蛋白溶液稀释为20 µg/mL、体积为100 µL,分别放在Regent Rack1试管架的A1、A2、A3、A4;另取一管加入200 µL NaOH(50 mM sodium hydroxide)作为再生液,放置在B1,将试管架放置于样品舱(见图1)。点击工具条中Run,选择Manual Run,进入界面后在Flow rate中输入10 µL/mL(注:除动力学测定外,流速一般设为10 µL/min),选择偶联通道,本实验将把蛋白偶联在Fc2上,所以选择Flow path 2;在右上方的下拉菜单中选择试管架,本实验选用Regent Rack1,点击Start,在Save Results As对话框中,把文件保存为pH scouting,点击Save。点击注射命令图标(Sample injection,红色),选择Vail/Well position旁的下拉菜单,在样品位置图中点击A1位置,设定进样时间(Contact time)为120秒(注意对话框底部显示的最小样品体积,确认试管中的样品体积满足要求),点击“OK”;点击再生命令图标(Regeneration injection,蓝色),在Vail/Well position选择B1位置,进样时间为30秒,点击“OK”。重复点击进样和再生命令,分别进样A2、A3和A4,时间为120秒,每次进样后再生30秒,所有命令都完成后,点击结束命令图标(Stop run),上述操作详见图3A表示样品放置位置,3B为上样顺序(pH5.5、5.0、4.5、4.0)、时间和再生时间。

图3 HSA蛋白偶联pH值的筛选。 A. 样品放置 B. 上样顺序(pH 5.5~4.0)、进样时间和再生
- 偶联HSA蛋白
偶联是指将蛋白固定到芯片上的过程,针对不同的芯片和蛋白,偶联方法各不相同。实验采用的芯片类型和偶联方式由多种因素决定,如配体蛋白的分子量、纯度、标签类型、蛋白母液成分、分析物分子量大小等。如果配体蛋白和分析物分子量差异非常大时,建议使用氨基偶联方法将配体蛋白偶联至CM5芯片;当偶联的配体蛋白不稳定或纯度稍低或偶联量要求不高时,可根据标签类型采用相应的捕获方法,如配体有His标签,先将anti-His蛋白偶联至CM5芯片表面,然后捕获带有His标签的蛋白;如果配体有Fc标签,可以采用Protein A、Protein G芯片;分析物与配体结合后再生困难的情况时也可以采用捕获法。本实验将采用最常用的氨基偶联法进行蛋白偶联。
在偶联时,实际偶联量建议接近理论偶量。蛋白偶联量RL的计算:Rmax = MW analyte/MW ligand *RL*Sm,其中Rmax = 100RU,Sm = 1,理论偶联量 = 1.5RL,Rmax、RL 的值都用Resonance Unit(RU)表示,MW analyte表示小分子化合物法华林的分子量308.33Da,MWligand表示HSA蛋白的分子量66,471.53Da。本实验计算所得RL = 21,558.6,1.5RL = 32,337.9。
先将分装冻存的100 µL EDC和NHS解冻并混匀,然后放置在Regent Rack1试管架的A1、将配制好的50 µg/mL的HSA蛋白放置在B1、将1.0 M(pH8.5)乙醇胺放置在C1,将试管架放置于样品舱。点击工具条中的Run,选择Manual Run,点击注射命令图标(Sample injection,红色),选择Vail/Well position旁的下拉菜单,在样品位置图中点击A1位置,设定Contact time(活化芯片表面时间),使芯片表面发生酯化;点击注射命令,选择B1,设定Contact time(蛋白偶联时间),与蛋白的氨基发生反应;点击注射命令,选择C1,设定Contact time(封闭时间),用乙醇胺封闭芯片上多余的有活性羧基,所有命令都完成后,点击结束命令图标(Stop run),具体操作如图4所示。(注意:偶联时间的长短需要根据所偶联蛋白的浓度以及理论偶联量决定,封闭时间一般和使用EDC、NHS活化芯片表面的时间一致。)

图4 HSA蛋白偶联过程。 A. 偶联试剂和样品放置; B. EDC/NHS活化、HSA偶联、乙醇胺封闭进样顺序和时间
- 小分子化合物Warfarin与HSA蛋白相互作用时测试条件摸索
该实验步骤的目的有:(1)确定化合物与蛋白是否有特异性结合;(2)分析化合物与芯片表面是否存在非特异性结合;(3)化合物选择多少浓度进行动力学分析;(4)结合时间和解离时间分别设置多少秒。
使用100%的DMSO将Warfarin配制成50 mM母液;用上样缓冲液HBS-EP+稀释Warfarin样品至50 µM、5 µM、0.5 µM,点击图标(Eject rack tray)退出试管架,分别将0.5 µM、5 µM、50 µM的样品放在A1、A2、A3,将上样缓冲液HBS-EP+放在B1。将试管架放回样品舱,点击sample injection图标,选择A1样品,进样时间设为30 s,点击ok;点击wait图标,设为30 s,点击ok;点击Regeneration injection图标,选择B1,进样时间设为30 s,点击OK。另外两个样品重复上述操作。 - 小分子化合物Warfarin的多循环动力学分析
经过上述摸索进样条件,初步了解到HSA和Warfarin结合解离性质以及亲和力大小。用上样缓冲液HBS-EP+稀释Warfarin样品,使其浓度分别为50、25、12.5、6.25、3.125、1.5625、0.78125、0 µM。应用向导程序设置多循环实验,进行动力学测试。打开New Wizard template,在 Assay中选择Kinetics/Affinity,点击New;在Injection Sequence 对话框中,Flow path选择2–1,Chip type选择CM5,点击Next,进入下一步;在Setup对话框中,设置Startup。在样品上样前,一般设置Startup循环,使系统和基线稳定,一般用上样缓冲液,在Solution中输入HBS-EP+,Number of Cycles设为3(通常为3–5次),点击Next,进入下一步;在Sample中,Contact time是指分析物的进样时间,本次实验为30 s,亲和力实验的Flow rate必须大于30 µL/min,此处设为30 µL/min;Dissociation time是指解离时间,需根据样品的情况设置,此处设为30 s;在Regeneration中,选用上述摸索的再生试剂,本实验根据解离情况选择使用HBS-EP+,Contact time设为30 s,Flow rate设为30 µL/min。在Samples对话框中填写样品名称和浓度信息,如果是同一样品,即使浓度不同,其名称也需保持一致。设置Warfarin浓度,分别为0、0.78125、1.5625、3.125、6.25、12.5、25、50 µM,点击Next进入下一步,去掉prime before run前的打勾,点击Cycle run list,确认实验运行中所有的循环是否正确,点击Next,进入下一步,将准备好的样品放置于样品架中的指定位置(图5),点击Next,检查各孔放置是否正确,点击Start,保存文件Kinetics data.blr,实验程序开始运行。
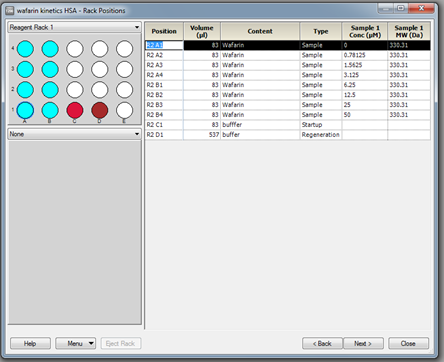
图5 HSA蛋白与Warfarin的多循环动力学样品的放置。
- 数据拟合
完成多循环动力学进样后,使用Biacore Evaluation Software 3.2对实验结果进行分析,主要步骤如图6所示,选择Kinetics/Affinty下的Surface bound选项,进入分析界面,在Select Curves界面中选取要分析的数据;在Select Data界面中,所有曲线都需扣减掉零浓度曲线,如果不需要分析传感图上的部分数据段,可点击鼠标右键,使用Remove selection 功能将其去除;界面的右下方有两个模式:Affinity和Kinetics,如果传感图是快上快下型(fast-on、fast-off,常见于小分子分析物),请选择Affinity,应用稳态模型Steady State进行分析;如果传感图呈动力学性质,选择Kinetics进行分析。本实验中,选择Kinetics,结合模式选择1:1,点击fit对数据进行拟合,获得结合速率常数(Ka)、解离速率常数(Kd)、亲和力(KD)。
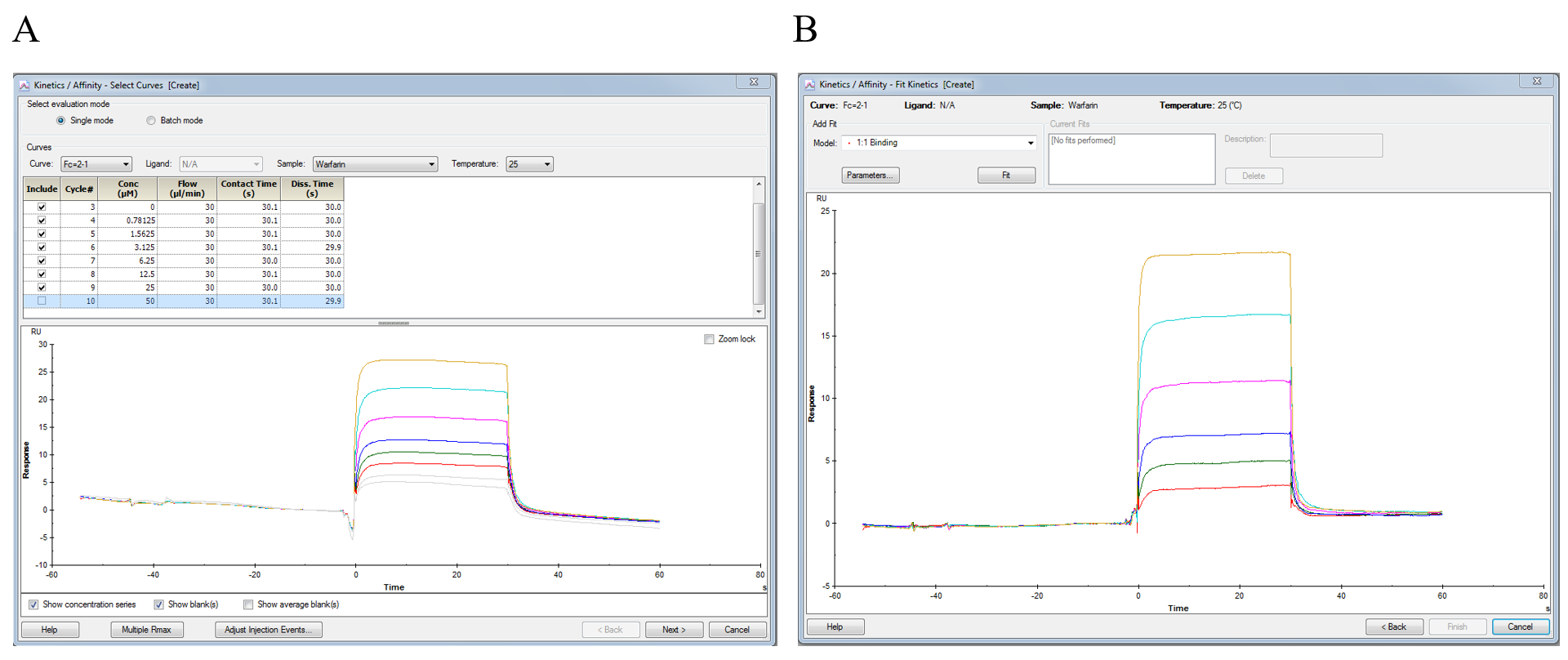
图6 HSA蛋白与Warfarin的多循环动力学拟合过程。 A.扣减零浓度前的传感图; B. 扣减零浓度后的传感图
实验结束后的操作
实验结束后,将芯片取出,装入维护芯片,将托盘左侧的上样缓冲液和右侧的上样针清洗缓冲液都换为超纯水,将右侧废液瓶倒掉、清洗后复位,然后执行desorb功能,先将desorb1 和desorb2放置于上样仓,点击Tools、More Tools、Desorb;结束后折行Prime功能,最后使仪器处于stand by待机状态(图7)。
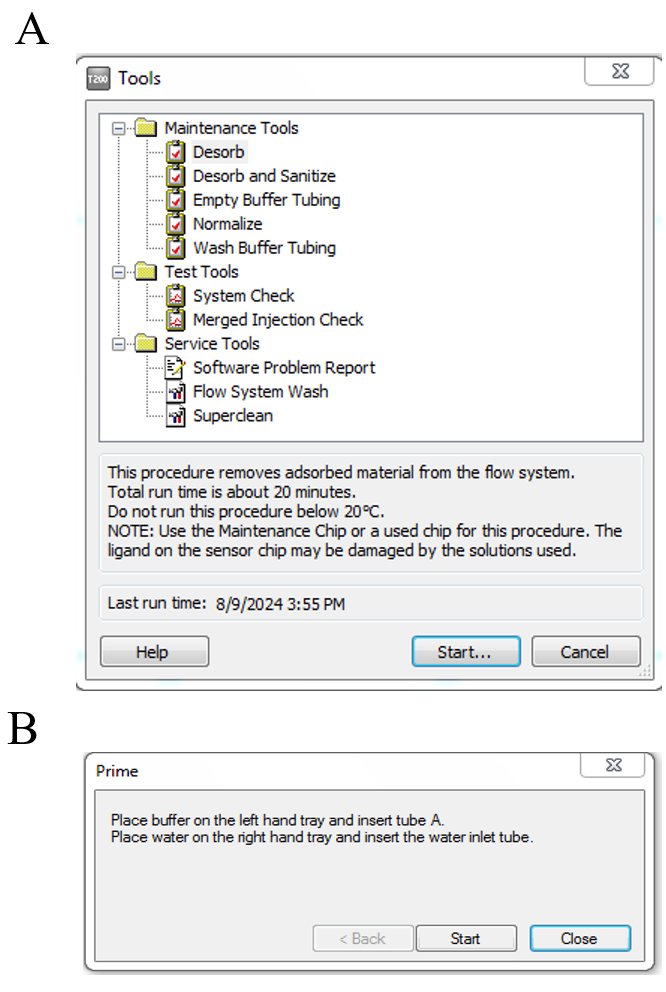
图7 实验操作结束后执行Desorb(A)和Prime(B)功能。
结果与数据分析
- HSA蛋白的pH筛选结果图
为保证HSA蛋白在Fc2通道上的偶联量,进行了不同pH值稀释的样品的预富集筛选,如图8所示,从左至右分别为pH5.5、5.0、4.5、4.0,结果显示,pH5.0条件下,蛋白在芯片上的预富集水平最高,下面实验将选择pH5.0的醋酸钠稀释蛋白进行偶联。
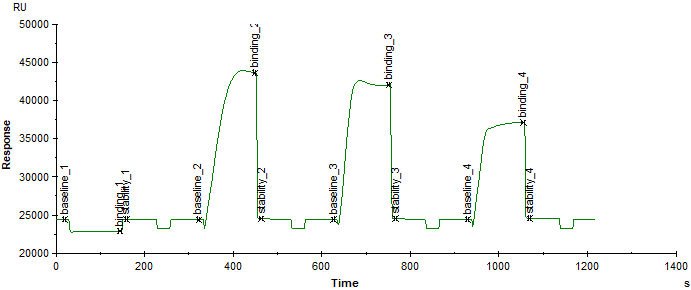
图8 HSA蛋白在pH 5.5、5.0、4.5、4.0条件下的预富集结果
- HSA蛋白的氨基偶联结果
采用手动偶联法,将HSA偶联至Fc2通道上,偶联过程如图9所示,先将各100 µL的EDC和NHS混匀后活化芯片表面720 s,然后先使用pH5.0的HSA偶联120 s,考虑到偶联量不足的情况下,又继续偶联60 s,最后用乙醇胺封闭芯片表面720 s,偶联过程结束。
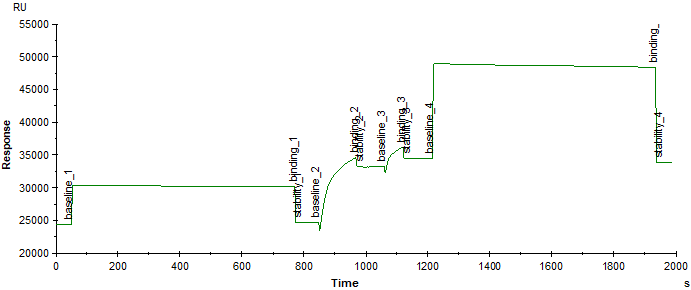
图9 HSA蛋白在pH 5.0条件下的偶联结果图
- HSA蛋白与Warfarin相互作用的分析结果
本实验使用Biacore T200进行了HSA蛋白与Warfarin相互作用的动力学分析,其结合、解离的传感图如图10所示,Warfarin流经芯片表面与HSA蛋白结合并很快达到饱和,进样结束后快速解离,其各浓度梯度间具有依赖性,取0、0.78125、1.5625、3.125、6.25、12.5、25µM,使用Kinetics,1:1 binding模式进行拟合分析,其结果如图11A所示,原始传感图与每个浓度拟合的黑色线条组成的图基本重合。数据拟合后图11B可观察到“Quality Control”中“Kinetic constants are within instrument specifications”指标显示绿色,表示动力学常数在仪器检测范围内,结果符合模型要求;“Kinetic constants appear to be uniquely determined” 指标显示绿色,表示动力学常数可以拟合出;“High bulk contributions(RI)found”指标显示黄色,表示存在一定的容积效应,但上述所有参数没有红色报警提示,说明获得的数据质量控质通过。图11C和表1所示,结合常数4.682E+4(1/Ms),解离常数为0.07800(1/s),亲和力KD为1.666E-6M。Rmax为3.422,远低于100RU;Chi2(RU)2值为0.294,小于Rmax的十分之一;U-Value值为12,小于15;Tc表示物质迁移系数,其值为9.423E+10,大于Ka值;上述都符合要求,说明该数据质量达标。
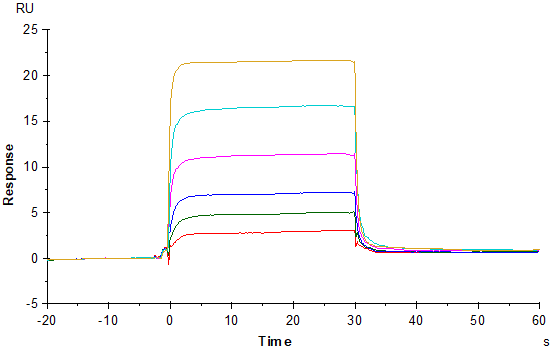
图10 HAS蛋白与Warfarin(0–25 µM)相互作用的信号传感图(拟合前)
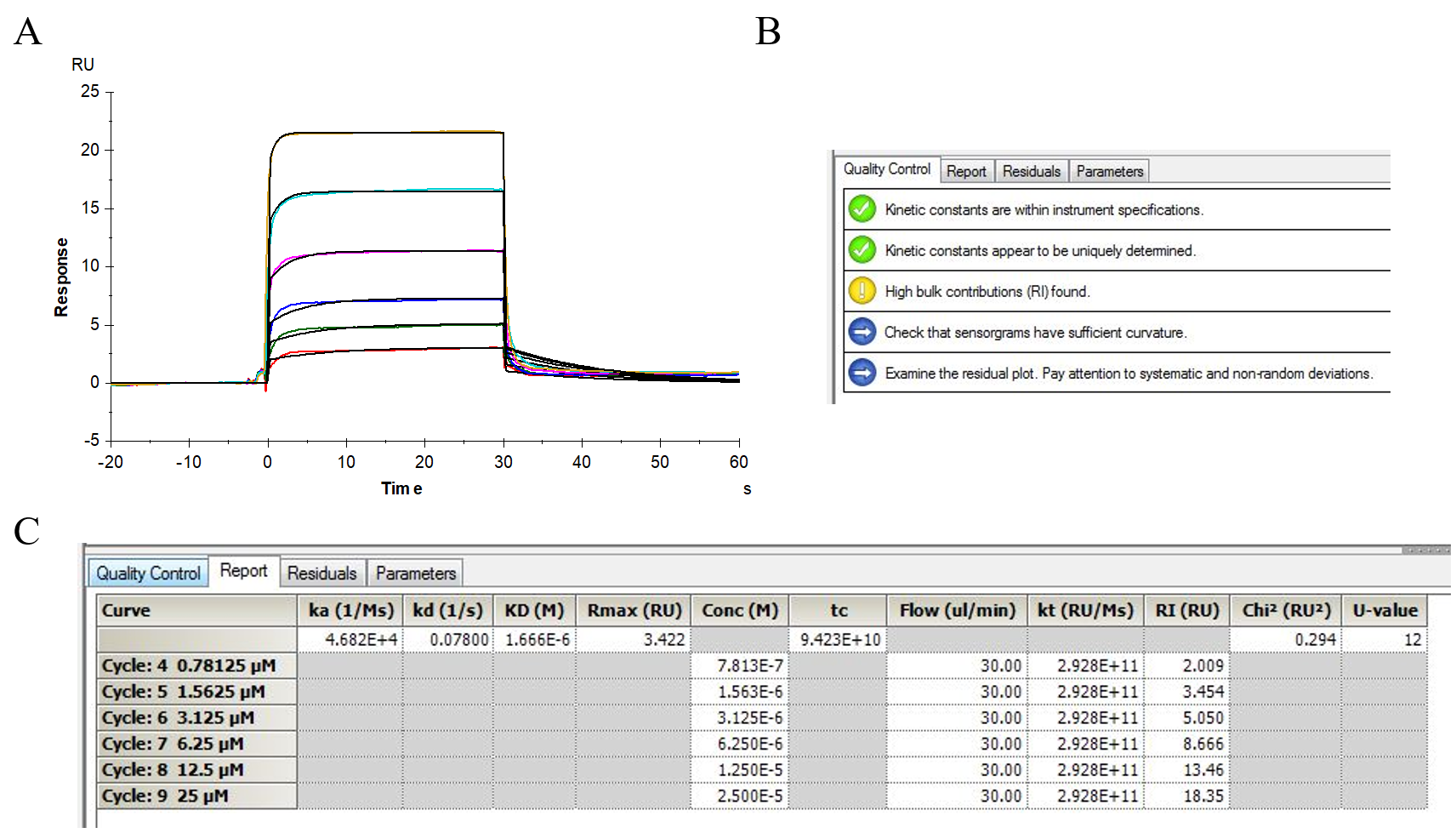
图11 HSA蛋白与Warfarin(0–25 µM)相互作用拟合后的传感图以及动力学参数。 A.HSA蛋白与Warfarin相互作用的信号传感图(Kinetics,1:1 binding拟合后);B.数据拟合后进行评估的Quality Control结果; C. 数据拟合后的Report结果
表1 HSA蛋白与Warfarin相互作用的动力学常数
| 样品 | 结合常数Ka(1/Ms) | 解离常数Kd(1/s) | 亲和力KD(M) |
| Warfarin | 4.682E+4 | 0.07800 | 1.666E-6 |
实验注意要点
- 实验前保证仪器状态正常
为保证仪器状态正常,需要执行System Check,确保各项参数都处于“Pass”状态(并不是每次实验前都需执行该程序,如果遇到异常情况时执行该功能判断仪器是否正常)。 - 如何选择合适的传感芯片?
根据实验需求选择合适的芯片,芯片种类如下:
(1)直接共价固定的芯片
CM5:最常用的传感芯片,大多数的分子间相互作用分析使用该芯片;
CM4: 芯片表面葡聚糖量低于CM5,可用于蛋白偶联水平要求较低的研究;
CM3: 芯片表面葡聚糖量低于CM4,大分子、病毒或细胞类的研究可用该芯片;
CM7: 芯片表面葡聚糖量低于CM5,可用于蛋白偶联水平要求较低的研究;
C1:适用于小型分子颗粒如细胞、病毒以及一些不需葡聚糖的研究的应用;
(2)间接捕获固定的芯片
NTA:适用于带有His标签的分子;
SA:生物素标记的分子,可使用不可逆性捕获的SA芯片;
Biotin CAP:生物素标记的分子,可使用可逆性捕获的CAP芯片;
Protein A、Protein G、Protein L:捕获抗体;
(3)疏水吸附固定的芯片
L1:研究脂质体分子时,可使用L1芯片直接捕获该分子进行研究(双分子层膜);
HPA:分析与膜系统相关的研究可以使用HPA芯片(单分子层膜);
(4)特殊定制的芯片
裸金Au芯片:芯片表面材料定制(材料、高分子)的芯片。 - 对所偶联的蛋白有什么要求?
蛋白尽量保存在PBS缓冲液中;纯度建议高于95%;浓度建议大于0.5 mg/mL,总量至少20 µg;保证蛋白具有活性;为避免干扰信号或影响偶联,建议缓冲液中不含甘油、蔗糖、甘露糖、甘露醇、海藻糖、咪唑等;如果使用氨基偶联法时,蛋白不能含有Tris、NaN3、甘氨酸等含伯氨基的成分。 - 对蛋白的偶联量有什么要求?
首先根据公式计算出RL,然后乘以1.5,得到蛋白的理论偶联量。如果在实际检测中,信号过低,可以补充偶联量,但如果已经达到芯片的饱和偶联量还达不到偶联要求,建议尝试使用CM7芯片。判断信号过低需要根据分子间相互作用的性质、分析物的浓度、产生的信号值大小以及结合前期实验结果或文献报道的亲和力值确定,如检测靶蛋白和小分子化合物之间的亲和力时,分析物浓度为50 μM时响应值的信号在0–5 RU之间,而文献报道或前期结果亲和力大约为0–10 μM之间,那么此时需要考虑偶联在芯片上的蛋白是否具有活性或分析物是否稳定。 - 实验时使用何种缓冲液?
大多数缓冲液可用,但需要根据分子间的结合性质、非特异性干扰、有无辅助因子参与等因素选择和配制合适的缓冲液,如参比通道非特异性吸附明显,需要使用HEPS-EP+缓冲液;如果蛋白和小分子结合时容积效应干扰大,需要在上样缓冲液(Running Buffer)中加微量的溶解小分子的溶剂;如果研究酶和底物的相互作用时,必要时在缓冲液中加辅助因子等。注意:上述缓冲液必须经0.22 µm滤膜过滤,并且建议现配现用。 - 对化合物进行浓度梯度稀释时,用什么缓冲液以及如何确定浓度的上限和下限?
使用上样缓冲液对化合物进行梯度稀释,其上限浓度根据实验前摸索的浓度设定,一般浓度范围为是亲和力值的0.1–100倍,然后倍半稀释至少5个浓度梯度,另外还需设置零浓度样品,所测得的亲和力值必须在浓度梯度范围内。 - 小分子化合物作为分析物时参比通道通有非特异性吸附,如何解决?
建议使用EP+缓冲液(含有表面活性剂P20)作为上样缓冲液,如果还存在,稍提高EP+ Buffer中P20的浓度,但不建议过高,否则会影响分子间相互作用。 - 为什么进样过程出现扣除参比通道的信号值呈现负值或降低趋势,如何解决?
将问题聚焦为分析物:缓冲液的pH是否偏低?分析物是否带正电荷?
(1)上样缓冲液pH过低,导致阳离子含量增高,容易非特异吸附在芯片表面,而未偶联蛋白的参比通道吸附性更强,完全掩盖了偶联靶标蛋白通道上的特异性信号,导致靶标蛋白通道信号扣除参比通道的信号为负值,如图12所示。解决方案:将缓冲液pH调整为7.4。

图12 酸性的缓冲体系导致Fc1非特异性强吸附而使Fc2–1呈现负值。
(2)分析物本身带正电荷,容易吸附在带有负电荷的芯片表面,并且与参比通道表面的吸附性更强,导致靶标蛋白通道信号扣除参比通道的信号呈现降低趋势,如图13所示。解决方案:可以将不与分析物结合的蛋白(Dummy Ligand,伪装配体)偶联至参比通道,以减少芯片表面携带的负电荷,降低非特异吸附。
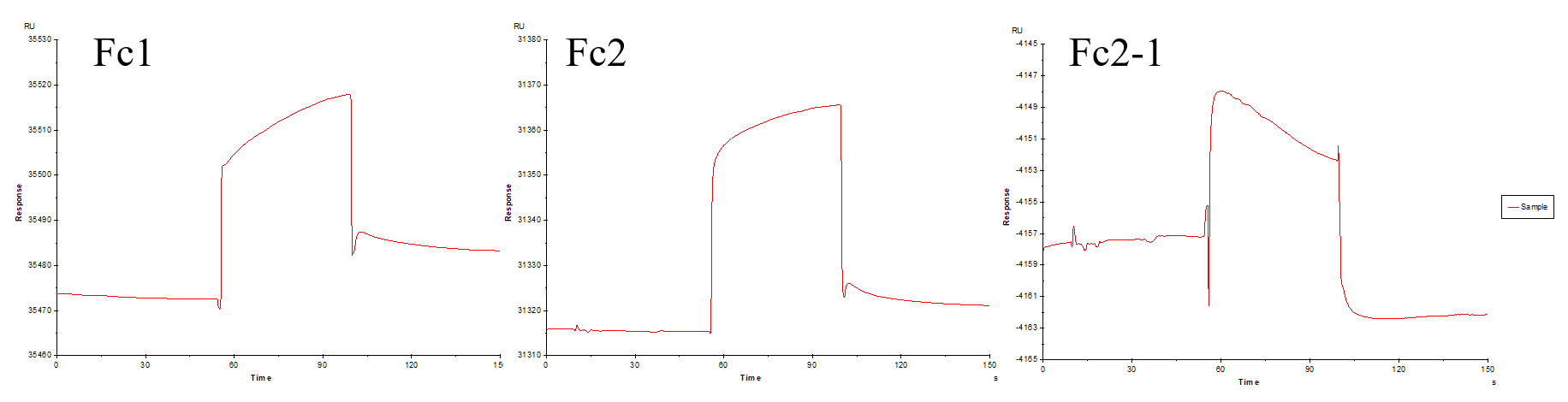
图13 分析物带正电荷导致Fc1非特异性强吸附而使Fc2–1呈现降低趋势。
- 蛋白或化合物不稳定时如何开展实验?
如果蛋白即使偶联在CM5芯片上也不稳定,可以通过捕获蛋白的方法进行分析;如果化合物不稳定,建议现配现用。 - 蛋白和分析物没有结合信号时如何解决?
这种情况一般要分析是蛋白的原因还是分析物的问题。判断是否是蛋白的问题,先通过设置阳性化合物,如果公认的阳性化合物和蛋白结合时没有信号,一般是蛋白的问题,要么是偶联量低或是蛋白没有活性;如果阳性化合物和蛋白结合有信号,那可能是分析物浓度低或不稳定的问题,采取调高浓度或现配现用的方式解决该问题。 - 在做蛋白与小分子相互作用分析时,如何解决化合物溶解性低、而与靶蛋白亲和力又弱的问题?
在进行蛋白与小分子相互作用分析时,会遇到化合物溶解性低且与靶蛋白亲和力又弱的情况。首先将化合物用100% DMSO溶解,将母液配制为10–20 mM,取1 µL加入到99 µL EP+ 缓冲液中,将900 µL EP+ 缓冲液加入上述试管中,用热吹风机吹管底,促进其溶解,然后以此为母液进行梯度稀释,为了溶解彻底,将每管涡旋并用吹风机也加热促溶,配制完成后迅速上样测定其亲和力,如果还存在较大的容积效应时,做溶剂校正曲线。 - 进样时,参比通道的RU值呈上升或下降趋势,为什么?
RU呈上升趋势时,说明分析物与参比通道有非特异性吸附;RU呈下降趋势时,说明缓冲液有干扰,如果对结果影响明显,需要更换缓冲体系。 - 为什么进样瞬间有一个或高或低的信号峰?
该现象说明样品与上样缓冲液折光率差异较大,要使所测数据准确、峰图漂亮,必须保证样品与上样缓冲液折光率一致,可以通过使用上样缓冲液配制和稀释样品;如果是DMSO配制的样品,可通过提高母液浓度而增加上样缓冲液的稀释样品倍数而解决。 - 进样结束后,一般选择什么样的试剂对芯片表面进行再生?
对于结合后容易解离的分子间相互作用,只需用上样缓冲液再生芯片表面即可;如果分子间结合后不易解离,需要逐步尝试从温和至强烈的不同pH的醋酸钠(3.0、2.5、2.0、1.5)再生,以及尝试使用NaOH(1–100 mM)、MgCl2(1–4 M)再生芯片表面。 - 进样结束后,解离到什么程度可对芯片表面进行再生?什么时候结束?
进样结束后,先使上样缓冲液流过芯片表面,使结合的化合物和蛋白自行解离,当RU降到最高浓度产生信号的10%后进行再生,信号基本回归到基线水平时结束再生。 - 进样结束后,分析物高浓度产生的信号不呈现梯度依赖性,而低浓度有,为什么?
这种现象说明该化合物溶解性低,高浓度时没有彻底溶解,产生的信号比实际低;低浓度时彻底溶解了,产生的信号与实际相符,因此呈现浓度依赖性。 - 蛋白与分子间进行多循环动力学分析时,浓度梯度与信号值为什么呈现反向依赖性?
这种情况并不是操作时将样品顺序放反了,而是因为化合物溶解性差,高浓度的化合物比低浓度下溶解的的分子少,产生的信号低,因此导致浓度梯度与信号值呈现反向依赖性。 - 对获得的数据进行拟合时,参比通道和零浓度的信号如何处理?
为了获得准确、客观的结果,拟合的数据需要采用双扣减,即扣减参比通道的信号和零浓度产生的信号。 - 蛋白与小分子化合物拟合时,为什么有的用kinetics拟合,有的用Affinity拟合?
当蛋白与小分子化合物结合后,解离较慢或稍快时,如果有解离曲线稍有弧度出现,用Kinetics拟合,可获得结合常数(Ka)、解离常数(Kd)、亲和力值(KD);当解离非常快时,即快上快下时,即稳态分析(Steady State),用Affinity拟合,此时没法计算出结合解离常数,只能求出亲和力。 - 如何评价数据拟合质量?
进行Kinetics拟合时,结合模式选择1:1,拟合后,首先确保Quality control没有红色报警项显示,说明拟合数据质控通过,然后观察Report栏中列出的结合速率常数(Ka)、解离速率常数(Kd)、亲和力(KD)、Rmax、Chi2(RU2)等重要数据,Rmax值要低于100RU,且Chi2(RU2)值越小越好,最大值要小于Rmax的十分之一;U-Value值小于15;Tc值表示物质迁移系数,其值越大越好,最小值要大于Ka值;如果上述都符合,说明该数据质量达标。
进行Affinity拟合时,因为是稳态分析即Steady State,结合解离性质呈现快上快下型,无法计算结合常数和解离常数,只能拟合出亲和力值,判断该值质控是否通过,可以观察亲和力值所对应的竖线是否落在各浓度形成的曲线内,并且Chi2(RU2)值越小越好,其上限值要小于Rmax(RU)的十分之一。 - 数据拟合时,所设的浓度梯度都需要采用吗?如何取舍?
实验设置浓度梯度时需要宽泛一些,以方便数据拟合时获得更为客观、准确的结果,根据拟合后Quality Control的评价、Report里的参数对其进行取舍,图14为本实验利用所有浓度(0~50 µM)进行拟合后的结果,发现与图11利用0~25 µM浓度进行拟合后的结果有差别,然后从Quality Control、Chi2(RU2)、U-Value等各项参数进行比较分析(见表2),结果表明采用0~25 µM浓度梯度的传感图拟合效果优于0–50µM的拟合效果,因此,HSA蛋白与Warfarin相互作用的动力学参数采用通过0~25 µM浓度梯度拟合的结果。
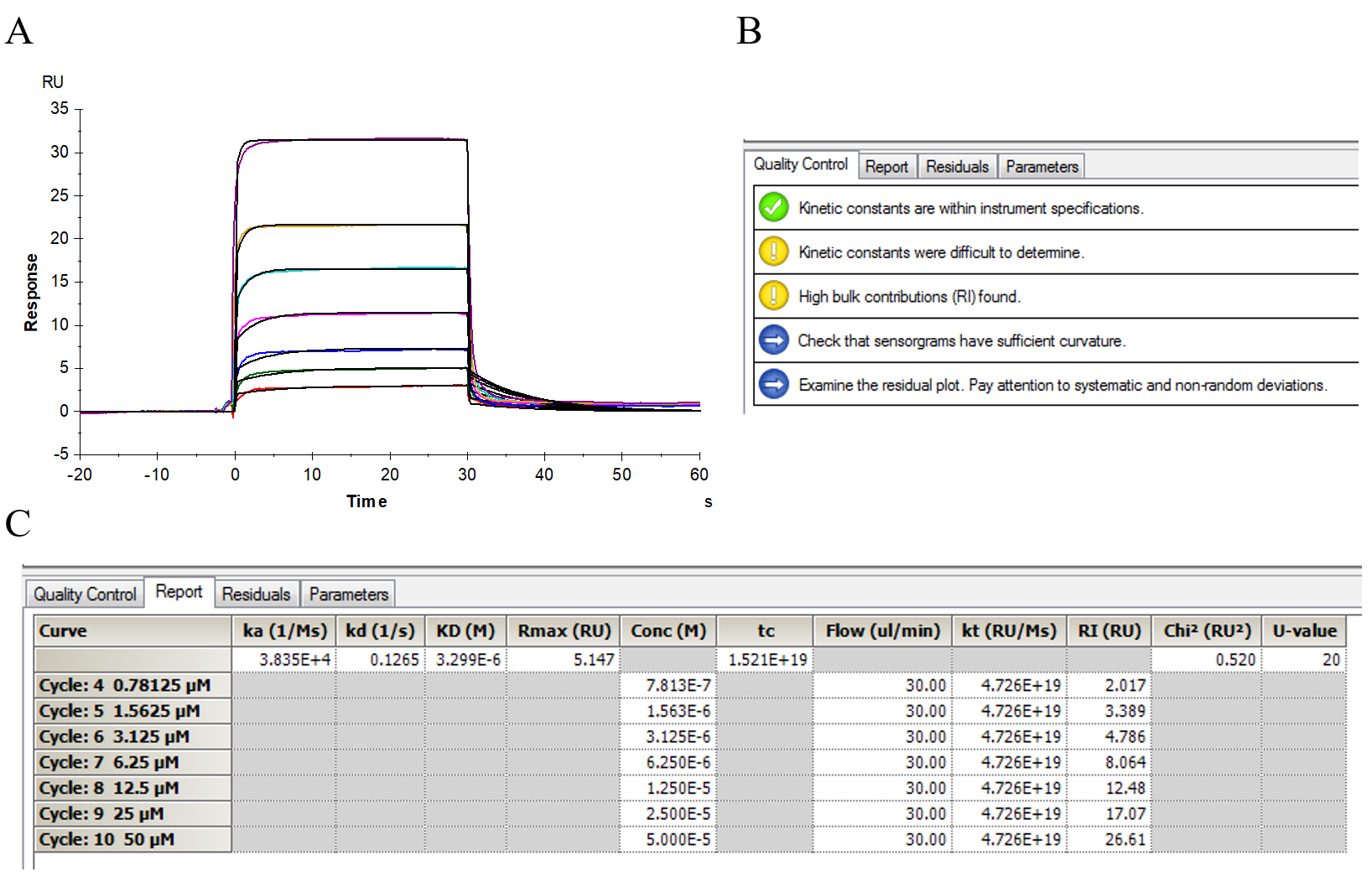
图14 HSA蛋白与Warfarin相互作用拟合后的传感图以及动力学参数。 A. HSA蛋白与Warfarin相互作用的传感图拟合后的结果; B.数据拟合后进行评估的Quality Control结果;C. 数据拟合后的Report结果
表2 HSA蛋白与Warfarin相互作用时不同浓度拟合后的数据比较
Warfarin
Concentration | Ka(1/Ms) | Kd(1/s) | KD(M) | Quality Control | Rmax | Chi2(RU2) | U-Value | tc |
| 0–25 µM | 4.682E+4 | 0.07800 | 1.666E-6 | GGY | 3.422 | 0.294 | 12 | 9.423E+10 |
| 0–50 µM | 3.835E+4 | 0.1265 | 3.299E-6 | GYY | 5.147 | 0.520 | 20 | 1.521E+19 |
注:G表示Green,Y表示Yellow
溶液配方
- HBS-EP+缓冲液
取50 mL购置的10× HBS-EP+缓冲液,加入到450 mL超纯水中,配制为1× HBS-EP+的缓冲液,建议现配现用。 - HBS-N缓冲液
取20 mL购置的10× HBS-N缓冲液,加入到180 mL超纯水中,配制为1× HBS-N的缓冲液,建议现配现用。 - 待测化合物的配制
将化合物用100% DMSO溶解,用1× HBS-EP+的缓冲液稀释至所需浓度。
致谢
本实验数据由复旦大学药学院仪器测试中心的Biacore T200设备产生,是为复旦大学药学院药剂学系蒋晨教授的文章2建立的分析测试方法。本论文只用来提供Biacore方法学参考,不涉及利益冲突,在此表示感谢!涉及的问题解决方案是本作者在该领域内方法学的拓展和延伸。
参考文献
- BiacoreTM T200 Instrument Handbook, GE Healthcare Life Science, USA.
- Zhang, Y., Guo, Z., Cao, Z., Zhou, W., Zhang, Y., Chen, Q., Lu, Y., Chen, X., Guo, Q., Li, C., et al. (2018). Endogenous albumin-mediated delivery of redox-responsive paclitaxel-loaded micelles for targeted cancer therapy. Biomaterials. 183: 243–257. https://doi.org/10.1016/j.biomaterials.2018.06.002
Copyright: © 2025 The Authors; exclusive licensee Bio-protocol LLC.
引用格式:曹忠莲, 杨萍. (2025). 利用SPR技术研究蛋白与小分子化合物相互作用的方法和解决方案. // Surface Plasmon Resonance (SPR) Protocol eBook.
Bio-101: e1011018. DOI:
10.21769/BioProtoc.1011018.
 How to cite:
How to cite: Cao, Z. L. and Yang, P. (2025). Strategy and Solution for the Studying of Protein-small Molecule Interaction by SPR Technology. // Surface Plasmon Resonance (SPR) Protocol eBook.
Bio-101: e1011018. DOI:
10.21769/BioProtoc.1011018.
