摘要:基于植物-微生物互作的生态修复技术是治理矿区土壤污染的重要方法,鉴于先锋植物根际微生物因其在促进植物生长中的巨大潜力,其微生物接种是一种维护植物健康生长与修复的重要方法。与商业化菌剂相比,从根际原位分离的土著微生物在定植效果和功能表现上更为优越,因此,快速判断具有分选潜力的根际样品尤为重要。同时,传统的菌株分离方法效率低下,目前仍缺乏获取根际菌株的高效方法。此外,单一菌株的定植效率和功能表现有限,而合成菌群(SynComs)能够更好地发挥协同作用。因而本文提出了一种结合样品识别、高效分离、稳定合成及植物促生验证的实验方法,用于矿区先锋植物根际微生物的快速分离与群落合成,为矿区生态修复提供技术支持。
关键词: 重金属, 植物内生菌, 高效分离, 稳定合成, 功能验证
研究背景
在矿山生态修复过程中,植物面临金属胁迫和寡营养等挑战,导致生态修复效果不佳。而先锋植物微生物组通过固氮、溶磷和分泌铁载体等机制,促进植物生长和营养吸收,并通过螯合或转化金属,降低重金属毒性,增强植物耐受性[1]。因此,根际微生物接种已成为促进植物修复的重要方法[2]。尽管商业菌株的应用已相当普遍,但其在根际中的定植和功能发挥受到环境显著影响[3],且由于缺乏对多样宿主的适应性,商业菌株的功效往往不够可靠,导致预期效果不佳[4]。而根系土著微生物更能适应当地环境,具有更好的定植效果和功能表现[5]。因此,快速筛选适宜分选的根际样品,并原位分离具有促生潜力的微生物至关重要。目前,常用传统平板划线分离方法分离目标功能菌株,但这种方法繁琐且周期长,分离效果差[6]。相比之下,通过极限稀释结合96孔培养板的分离能够显著提高分离效率,可以在短时间内获得大量纯种菌株,并结合基因测序,可快速获取微生物的遗传信息及代谢功能。考虑到单一微生物的功能有限难以适应复杂的环境,由单菌合成得到的低复杂性和高可控性的SynCom的研究也越来越受到关注[7]。例如,土著SynCom可通过多种植物促生机制有效提高植物的生产力和抵御胁迫的能力[8,9]。这些SynCom还能与原生土壤中的植物宿主共同进化,尤其是在矿区等营养有限的环境条件下,土著SynCom对土壤功能和植物发育具有持久的积极影响[10]。因此,如何选择功能互补且稳定的菌群成员并构建有效的SynCom成为当前研究的重点。基于此,本研究提出了一种用于识别、分离、高效合成矿区先锋植物根际促生菌群的方法。
主要流程
首先快速判断和筛选出具有分选潜力的先锋植物根际样品。随后,采用极限稀释结合96孔培养板的方法对这些样品中的根际微生物进行高效分离,并通过基因测序获取其遗传信息和代谢功能。最后,基于所筛选出的功能菌株,构建低复杂性且高可控性的合成菌群,并通过植物促生实验验证其在矿区生态修复中的潜力(图1)。
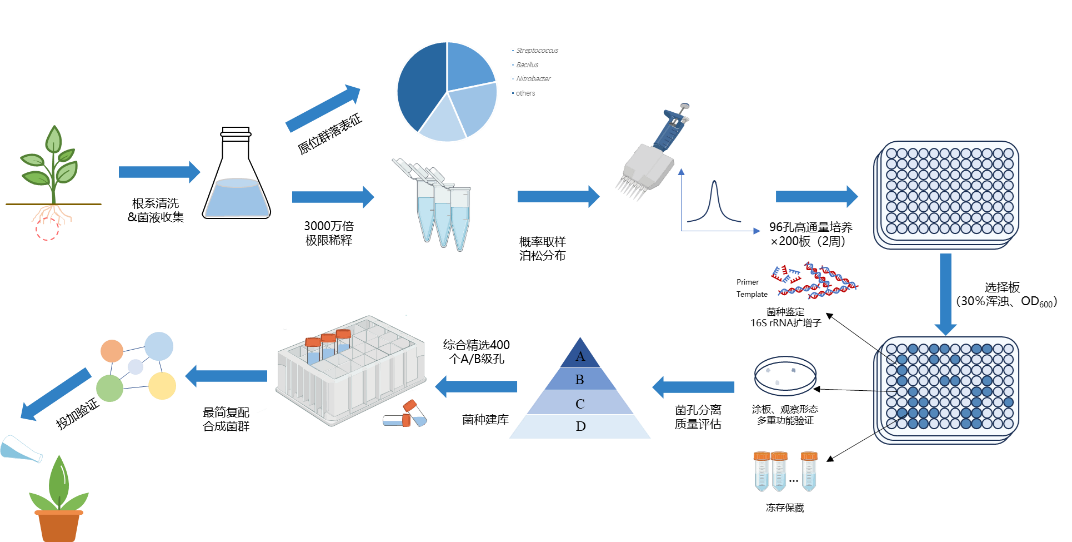
图 1. 本文工作流程示意图
材料与试剂
- 移液器枪头
- EP管
- 50 mL离心管
- 96孔细胞培养板(比克曼)
- Parafilm封口膜 (比克曼)
- 0.22 μm 无菌过滤膜(比克曼)
- PCR板 (比克曼)
- 营养土壤
- 珍珠岩
- 蛭石
- 营养钵
- 次氯酸钠(上海国药,分析纯)
- 氯化镁(上海国药,分析纯)
- 酒石酸锑钾(上海国药,分析纯)
- 70%乙醇溶液
- 琼脂粉(上海国药,分析纯)
- 无菌去离子水
- 磷酸缓冲液 (biosharp)
- FeCl3(上海国药,分析纯)
- 浓H2SO4(上海国药,分析纯)
- 吲哚乙酸(上海国药,分析纯)
- 拟南芥(野生型 col-0)
- 甘油(上海国药,分析纯)
- 液氮
- 大肠杆菌DH5α(Thermo Scientific)
- Loading Buffer(Biosharp BL532A)
- 碱裂解Buffer I(25 mM NaOH and 0.2 mM Na2—EDTA,pH = 12)(Biosharp BL532A)
- 中和缓冲液Buffer II(40 mM Tris-HCI,pH=7)(Biosharp BL532A)
- 2.5%有效氯浓度的次氯酸钠溶液(见溶液配方)
- 1/2 MS 固体培养基(见溶液配方)(比克曼)
- 1/10 TSB 液体培养基(见溶液配方)(青岛海博)
- LB 液体培养基(见溶液配方)
- 无机磷培养基(见溶液配方)(青岛海博)
- Ashby无氮培养基(见溶液配方)(青岛海博)
- CAS培养基(见溶液配方)(青岛海博)
- R2A固体培养基(见溶液配方)(青岛海博)
- SV Gel and PCR Clean-up System试剂盒
- Quant-iTTM PicoGreenTM dsDNA Assay Kit分析试剂盒
- DNeasy Powersole试剂盒(Qiagen,Dresden,德国)
仪器设备
- 生物安全柜/超净工作台(Thermo Fisher, 1300 Series A2)
- 高压蒸汽灭菌锅(力辰,DGL-35G)
- 可见分光光度计(北京普析,T400F)
- 电子天平(力辰,LC-FA2004)
- 高速离心机(Eppendorf, 5810r)
- 八通道移液器(Thermo, Scientific)
- 三角锥型瓶(蜀牛)
- 微孔板离心机(迈克尔,MK-30)
- 恒温培养摇床(一恒,THZ)
- 超低温冰箱(Thermo, 906-Ults)
- PCR仪(Bio-Rad, CFX96)
- 凝胶电泳仪(伯兰特,DYCP-31DN)
实验步骤
一、分选潜力样品的快速判别
- 从矿区自然土壤中采集植物新鲜根系样品,使用冰袋运回实验室。为了保证样品中微生物的活性,样品应尽量在4小时内运达实验室进行下一步分析。采集先锋植物(如芒草)的新鲜根系样品约10 g,首先用自来水将肉眼可见的土壤颗粒洗掉。接着将根系转移至新的50 mL离心管,管中加入30 mL磷酸缓冲液,在室温条件下,放置在平板摇床上用180 rpm的转速清洗15 min,重复清洗3遍。最后,用无菌滤纸吸取根系残留液体。
- 将根系研磨成匀浆。将清洗并吸水后的根系组织用无菌剪刀尽可能剪成小段,并混合均匀。称取2 g的根系组织放入1.5 mL的无菌离心管。在生物安全柜中,向离心管中加入200 μL灭菌的10 mM的氯化镁溶液,用无菌研磨棒研磨根系直至成匀浆状,取混匀的根系组织用于检测根系微生物组。
- DNA提取和扩增
使用DNeasy Powersole试剂盒提取内生菌DNA。将基因组DNA作为模板,扩增其V5V7高变区。以799F为正向引物,以1193R为反向引物进行PCR扩增;已知菌液大肠杆菌DH5α为模板的阳性对照,灭菌去离子水为模板的阴性对照;PCR扩增产物进行琼脂糖凝胶电泳检测,配制1.2%的琼脂糖凝胶,电压设置120 V,电泳约20–40 min。阳性对照目的条带位置正确,阴性对照无条带,表明PCR扩增合格。切取16S rRNA基因的PCR扩增片段(约500 bp)以及该片段上下2 mm凝胶内的所有片段,然后将胶块装入2 mL无菌离心管,用SV Gel and PCR Clean-up System试剂盒进行切胶回收,得到各孔细菌的16SrRNA基因的PCR纯化样本。DNA浓度由Quant-iTTMPicoGreenTM dsDNA Assay Kit分析试剂盒(Invitrogen公司)测定,并以等量DNA的标准混合细菌DNA样本,得到测序鉴定文库。 - 测序和生信分析:对鉴定文库在Hiseq2500平台上进行测序,得到根系细菌的非冗余序列,每个序列包含一个barcode和16S rRNA基因V5-V7序列。根据barcode对序列进行质量过滤和样本拆分,通过UNOISE算法,去除嵌合体和低丰度序列(读取计数<8)后定义identity = 100%的序列为ASV。将培养细菌16SrRNA基因V5-V7序列与Silva (https://www.arb-silva.de/)数据库中的细菌16S rRNA基因的V5-V7序列进行比较,采用SINTAX算法对细菌ASV进行分类注释。对其进行PICRUSt2基因预测,功能基因参考KEGG数据库,以植物促生相关功能基因为筛选阈值,具体可根据实验目的进行调整。
- 当根际微生物群落的PICRUSt2基因预测结果显示具有显著表达的与植物促生相关的功能基因(如ipdC、acrA、mbtH、nifH等)和与特定污染物抗性相关的基因(如aioA、arsA、czcD等)时,可判定该根系样品具有分选潜力,适合作为进一步分离与研究的候选样品。
推荐例:
砷/锑污染土壤:锑抗性相关基因
aioA(K08356),
arsA(K01551),
arsB(K03325),
arsC(K03741)。
镉污染土壤:
czcD(K16264),
zipB(K16267),
cmtR(K21885),
cadC(K21903)
植物促生:
ipdC(K04103),
acrA(K03585),
mbtH(K05375),
nifH(K02588)
二、根系微生物的高效分选与功能验证
- 根系微生物高效分选(图2)
- 重复上述根系分离步骤,将得到的根系匀浆液转至含有25 mL的10 mM氯化镁的50 mL管中,混匀,室温下静置15 min。在生物安全柜中,例如将菌液按照10万倍,100万倍,1,000万倍,1亿倍稀释倍数加入到含有0.1 mM Sb3+ 的灭菌10% TSB溶液的4个瓶中。摇匀每瓶稀释液,将部分稀释液倒入无菌培养皿中,用排枪吸取液体移入96孔细胞培养板的每个孔中,每孔160 μL,每瓶稀释液转移3个细胞培养板,未添加根系匀浆的灭菌10%TSB溶液转移3个细胞培养板作为阴性对照。用Parafilm将每一个细胞培养板封口,反复仔细多刮表面多次。移液时应尽量避免液体溅到孔外,否则容易造成污染。避光一周后寻找与30%浑浊度最接近的稀释倍数作为最适稀释浓度(ODC),重新按照上述步骤研磨根系匀浆,并分别按1/3× ODC、ODC和3× ODC稀释浓度用0.1 mM Sb3+ 的10%TSB溶液配制稀释液。将稀释液转移至96孔板,放置在室温下孵育2周。二周后观察96孔细胞培养板中细菌的生长情况,保留约30%的孔呈现肉眼可见的浑浊状态的培养板。用微孔板离心机将板内液体离心至管底再揭开PCR封板膜以避免污染。用移液枪从培养板孔中吸取20 μL样品移入灭菌EP管中,存储在-20 °C冰箱用于后期细菌鉴定。每孔剩余样品中转移新的灭菌EP管并添加140 μL的80%灭菌甘油,使用液氮速冻后存储在-80 °C冰箱。
- 菌孔DNA的提取鉴定(本步骤也可以经后续功能鉴定后再选择所需目的菌孔进行鉴定):采用碱解法提取培养细菌的DNA。向20 µL样品移入灭菌EP管中加入20 μL碱性裂解液。将上述体系用排枪吹打混匀后用扣盖,在水浴锅(95 °C,30min)裂解菌体。冷却降温后,向每孔中加入20 µL中和缓冲液,混匀并离心,存储于-20 °C冰箱。再参考上述步骤对DNA进行扩增和生信分析。
- 取-80 °C冰箱冻存菌株在LB固体培养基中活化后,挑取单菌落转接至20 mL LB液体培养基中,置于28 °C、转速180 r/min条件下的恒温培养摇床中过夜培养。将富集培养的菌液,使用可见光分光光度计调整菌液浓度至OD600 = 0.5后,用于下一步接种。
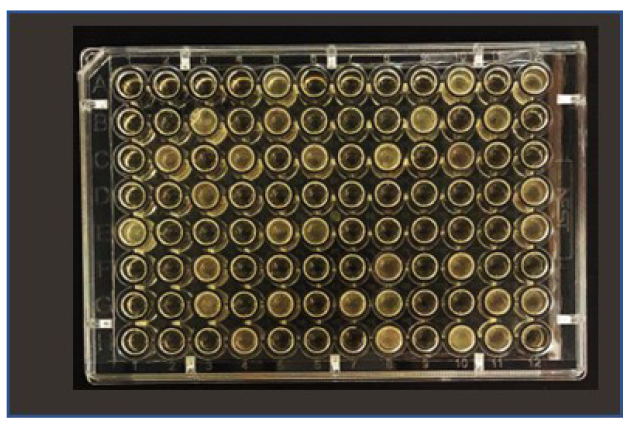
图 2. 菌群高效分离示意图
- 菌群常见促生功能验证(根据目的选择)(图3):
- 溶磷功能研究:取1 μL OD600 = 0.5的菌液接种于无机磷培养基上,每株重复3次,37 °C培养7 d。观察有无溶磷圈产生,以接种1 μL的空白LB培养基为对照组。
- 固氮功能研究:取1 μL OD600 = 0.5的菌液接种于Ashby无氮培养基7 d,观察其生长情况和菌落周围是否出现透明圈,如出现透明圈则该菌株具有固氮能力,以接种1 μL的空白LB培养基为对照组。
- 产铁载体功能研究:取1 μL OD600 = 0.5的菌液接种于CAS培养基,37 d,观察其生长情况,观察菌落周围是否出现黄色圈,如出现黄色圈则该菌株具有产铁载体功能,以接种1 μL 的空白LB培养基为对照组。
- 产IAA功能定性测定:供试菌株接种于100 mL LB液体培养基(加入0.5 g/L色氨酸)。置于30 °C摇床,180 r/min 4 d。取菌悬液10,000 r/min离心5 min,取上清液1 mL于10 mL,同时加入4 mL比色液(30 mL浓H2SO4,1.5 mL 0.5 mol/L比色液 FeCl3,50 mL蒸馏水),标准对照在比色液中加入1 mL 50 mg/L的吲哚乙酸标准品,空白对照加入1 mL培养基和4 mL比色液。所有EP管于室温避光放置30 min后观察,颜色变红者表示能够分泌IAA。
- 金属耐受测定:使用的培养基为LB液体培养基,加入重金属溶液(可根据需要设置浓度梯度),设置3个平行组以及1个空白对照(不接种菌),每组配制250 mL培养液,进行高温灭菌(121 °C,20 min),在超净工作台上冷却至适宜温度后,接入1%的菌液,锥形瓶均放到30 °C、150 rpm的摇床中培养,实验持续3天。每间隔12或24小时用可见光分光光度计测定其OD600并绘制生长曲线。
- 如所选重金属为变价金属(砷锑),可同步每间隔12或24小时测定一次氧化/还原情况,绘制氧化/还原曲线。曲线测定时,先将样品在50,00 rpm下离心5 min,取上清液,使用原子荧光测定其中砷锑(III)的浓度,绘制浓度曲线。
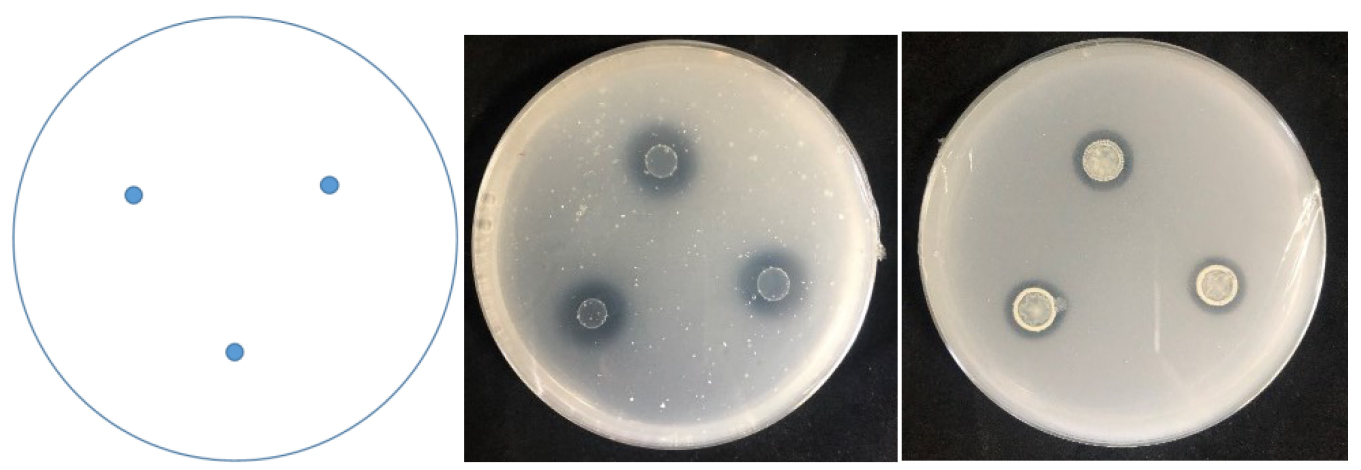
图 3. 功能验证示意图(溶磷,固氮为例)
三、生态稳态构建及植物促生功能验证
- 菌株互作
- 平板对峙实验:使用可见光可见分光光度计测定菌液浓度,将浓度调节至OD600 = 0.5;吸取7次1 μL稀释液依次接种至R2A固体培养基的方形培养皿的两侧对角线,接种点间逐渐靠近呈现一个“V”型。封口膜密封后,20 °C孵育7天,在垂直于“V”型的直线上观察菌落生长情况,并对菌落直径进行测量。选择互利共生的菌株构建合成菌群进行进一步实验。本方法用来探究挥发性物质对菌群的影响(图4)。
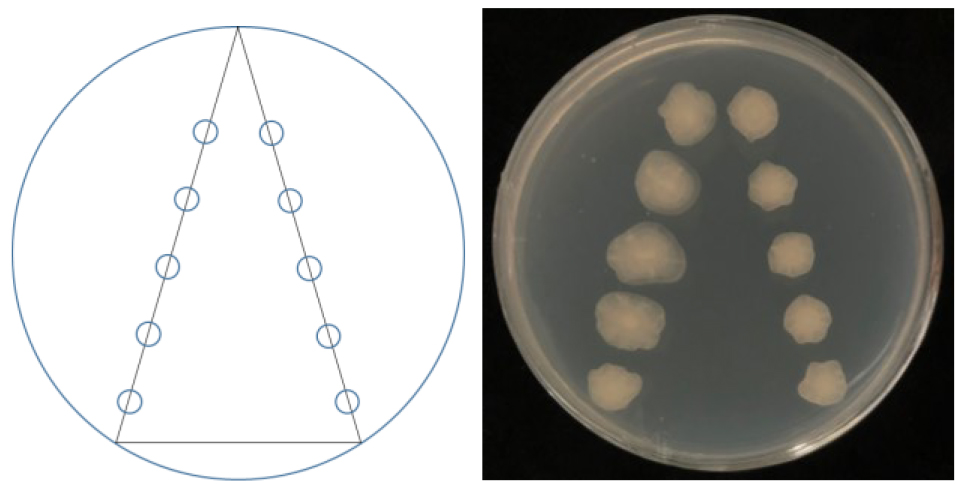
图 4. 平板对峙示意图
- 液体互作实验:将两种细菌分别接种到20毫升的LB培养基中,并在37 °C、180 r/min的条件下孵育48小时,直至达到稳定期。然后,将细菌培养液以8,000 r/min离心10分钟,收集上清液(SM),并通过0.22 μm的无菌滤膜过滤。将每种细菌的细菌悬液调整到相同的光密度(OD600 = 0.1),确保细菌数量相同,并以1%的体积比接种到另一种细菌的SM中,在37 °C下孵育48小时。作为对照,将每种调整好的细菌悬液分别接种到新鲜的LB培养基中。孵育结束后,测量所有培养液的OD600值,表示细菌的生长量。结果表示为细菌在不同SM中的OD600值除以同一种细菌在新鲜LB培养基中的OD600值(即OD600 SM/新鲜)。本方法用来探究非挥发性代谢物质对菌群的影响。
合成菌群的构建则根据以上结果进行综合判断,按照菌OD600 = 0.5,体积比为1:1的结果来进行构建。
- 植物促生验证(图5)
- 挑选一批籽粒饱满的野生型拟南芥种子,置于无菌的锥形瓶中,加入75%的酒精浸泡5 min,然后用无菌水清洗4次;使用2% NaClO溶液浸润浸泡10 min,期间不断摇晃,随后使用无菌水清洗6次,将种子表面的NaClO清洗干净。将拟南芥种子点在1/2 MS的隔板培养基上,置于4 °C的冰箱春化48 h(以上步骤均在超净工作台中进行)。将无菌培养体系置于25 °C,16 h光照,25%湿度的培养间内培养7 d。将营养土壤、珍珠岩和蛭石按照体积比(3:1:1)混合均匀,在间隔24 h的高压121°C 20 min的灭菌条件下处理3次后烘干使用。将上述混合基质装入黑色营养钵后,取培养7天的拟南芥洗净根部残留培养基竖直放入基质中。置于25 °C,16 h光照,25%湿度的培养间内培养10 d(约4片真叶)后进行下一步处理。
- 将富集培养的菌液分别转移到50 mL的无菌离心管中,5,000 r/min、离心10 min后,于超净工作台中将其上清废弃;加入无菌水吸打均匀,置于上述离心条件再次离心,废弃上清(重复两次);加入无菌水重悬,使用可见光分光光度计调整菌液浓度至OD600 = 0.5后,按照菌液体积比基质重量为1:100的比例将菌液接种至营养钵内。若添加的微生物为混合菌群,将各成员按1:1等比例混合均匀后,再次测量并调整OD600至0.5,按比例添加至黑色营养钵内。可根据实验需要添加重金属,盐碱胁迫。继续置于25 °C,16 h光照,25%湿度的培养间内培养。

图 5. 合成菌群植物促生示意图
成功经验/失败经验
- 在含有胁迫的筛选培养基分选中,选择整体浑浊度低于30%的培养板。
- 可以利用96孔板进行所需功能筛选后测序,以减少非必要功能菌的获得。
- 合成菌群选择互利共生菌株(至少不拮抗),最好选择功能互补的菌株,能够充分利用生态位
溶液配方
- LB 液体培养基
蛋白胨10 g;酵母粉5 g;NaCl 8 g;蒸馏水1,000 mL;pH7.0–7.2。 - 1/10 TSB 液体培养基
胰酪胨17.0 g;氯化钠5.0 g;大豆木瓜蛋白酶水解物3.0 g;磷酸氢二钾2.5 g;葡萄糖2.5 g; - 无菌去离子水
去离子水经过121 °C高压灭菌15 min。 - 磷酸缓冲液
取磷酸二氢钾0.68 g,加0.1 mol/L氢氧化钠溶液29.1 mL,用水稀释至100 mL。 - 无机磷培养基
葡萄糖 10.0 g;硫酸铵 0.5 g;酵母浸粉 0.5 g;氯化钠 0.3 g;氯化钾0.3 g;硫酸镁0.3 g;硫酸亚铁0. 03 g;硫酸锰0.03 g;磷酸三钙5.0 g;pH值7.0-7.5。 - Ashby无氮培养基
磷酸二氢钾 0.2 g;硫酸镁0.2 g;氯化钠0.2 g;碳酸钙5.0 g;甘露醇10.0 g;硫酸钙 0.1 g;pH值7.0 ± 0.1。 - CAS培养基
铬天青S(CAS) 60.5 mg;十六烷基三甲基溴化铵(HDTMA) 72.9 mg;六水氯化铁 2.645 mg;二水磷酸二氢钠 295.25 mg;十二水磷酸氢二钠 1213.5 mg;氯化铵 125 mg;磷酸二氢钾 37.5 mg;氯化钠 62.5 mg;琼脂 9,000 mg;pH值6.8 ± 0.1。 - 1/2 MS 培养基
Murashige Skoog培养基(含维生素)干粉:2.2 g;蔗糖10 g;琼脂粉8 g; pH 值5.8。 - 2.5%有效氯浓度的NaClO溶液
100 mL 无菌去离子水加上 3 mL NaClO。 - 25 μL PCR扩增体系
1 μL模板DNA、0.25 μL Fast pfu DNA聚合酶、5 μL 5× PCR Buffer(Mg2+ Plus)、2 μL dNTPs、1 μL正向引物799F和1 μL反向引物1193R、14.75 μL ddH2O。- 799F:5’-AACMGGATTAGATACCCKG-3’
- 1193R:5’-ACGTCATCCCCACCTTCC-3’
灭菌要求:121 °C高压灭菌15 min,于超净台中分装,在4 °C条件下可存储数周。
PCR条件:DNA在98 °C下初始变性5 min,随后26个循环,每个循环在98 °C下变性30秒,在53°C下退火30 s,在72°C下延伸45 min,最后在72 °C下终延伸5 min,降温到16 °C保存PCR产物。
致谢
感谢湖南省自然科学基金杰出青年项目 (2024JJ2072)、国家自然科学基金面上基金项目 (42377033)、中南大学研究生自主探索创新项目(No. 2024ZZTS0321)对本工作的大力支持。
参考文献
- Singh, B. K., Trivedi, P., Egidi, E., Macdonald, C. A. and Delgado-Baquerizo, M. (2020). Crop microbiome and sustainable agriculture. Nat Rev Microbiol. 18(11): 601–602. https://doi.org/10.1038/s41579-020-00446-y
- Sharma, P. (2021). Efficiency of bacteria and bacterial assisted phytoremediation of heavy metals: An update. Bioresour Technol. 328: 124835. https://doi.org/10.1016/j.biortech.2021.124835
- Hartmann, M. and Six, J. (2022). Soil structure and microbiome functions in agroecosystems. Nat. Rev. Earth Environ. 4(1): 4–18. https://doi.org/10.1038/s43017-022-00366-w
- Kaminsky, L. M., Trexler, R. V., Malik, R. J., Hockett, K. L. and Bell, T. H. (2019). The Inherent Conflicts in Developing Soil Microbial Inoculants. Trends Biotechnol. 37(2): 140–151. https://doi.org/10.1016/j.tibtech.2018.11.011
- Liu, C., Jiang, M., Yuan, M. M., Wang, E., Bai, Y., Crowther, T. W., Zhou, J., Ma, Z., Zhang, L., Wang, Y., et al. (2023). Root microbiota confers rice resistance to aluminium toxicity and phosphorus deficiency in acidic soils. Nat Food. 4(10): 912–924. https://doi.org/10.1038/s43016-023-00848-0
- Lee, D. J., Show, K. Y. and Wang, A. (2013). Unconventional approaches to isolation and enrichment of functional microbial consortium – A review. Bioresour Technol. 136: 697–706. https://doi.org/10.1016/j.biortech.2013.02.075
- Zhou, Y., Liu, D., Li, F., Dong, Y., Jin, Z., Liao, Y., Li, X., Peng, S., Delgado-Baquerizo, M., Li, X., et al. (2024). Superiority of native soil core microbiomes in supporting plant growth. Nat Commun. 15(1): 6599. https://doi.org/10.1038/s41467-024-50685-3
- Niu, B., Paulson, J. N., Zheng, X. and Kolter, R. (2017). Simplified and representative bacterial community of maize roots. Proc Natl Acad Sci USA. 114(12): E2450–E2459. https://doi.org/10.1073/pnas.1616148114
- Qiao, Y., Wang, Z., Sun, H., Guo, H., Song, Y., Zhang, H., Ruan, Y., Xu, Q., Huang, Q., Shen, Q., et al. (2024). Synthetic community derived from grafted watermelon rhizosphere provides protection for ungrafted watermelon against Fusarium oxysporum via microbial synergistic effects. Microbiome. 12(1): 101. https://doi.org/10.1186/s40168-024-01814-z
- de Souza, R. S. C., Armanhi, J. S. L., Damasceno, N. d. B., Imperial, J. and Arruda, P. (2019). Genome Sequences of a Plant Beneficial Synthetic Bacterial Community Reveal Genetic Features for Successful Plant Colonization. Front Microbiol. 10: e01779. https://doi.org/10.3389/fmicb.2019.01779
Copyright: © 2024 The Authors; exclusive licensee Bio-protocol LLC.
引用格式:高寒冰, 何晓, 贾沃, 郭朝晖, 徐锐. (2024). 矿区先锋植物根内生促生菌的高效分离与群落稳定构建. // 微生物组实验手册.
Bio-101: e2405584. DOI:
10.21769/BioProtoc.2405584.
 How to cite:
How to cite: Gao, H. B., He, X., Jia, W., Guo, C. H. and Xu, R. (2024). The Efficient Isolation of Growth-Promoting Endophytic Bacteria from Pioneer Plants in Mining Areas and the Stable Construction of Microbial Communities. // Microbiome Protocols eBook.
Bio-101: e2405584. DOI:
10.21769/BioProtoc.2405584.
